Els animals marins de cos bla, com els nudibranquis, utilitzen compostos bioactius com a defensa contra depredadors. Anteriorment, en el nostre treball sobre el nudibranqui Doriopsilla fulva hem descobert que aquests compostos poden ser produïts per simbionts bacterians en lloc dels propis animals. Aquests bacteris simbiòtics representen nous llinatges bacterians no cultivables que produeixen molècules bioactives. El projecte Nudi-Symb cerca explorar més espècies de nudibranquis. Utilitzarem tècniques noves per a estudiar cèl·lules individuals bacterianes, seqüenciarem el genoma de nudibranqui i utilitzarem transcriptòmica per a entendre la interacció entre aquests animals i els seus bacteris simbiòtics, amb l'objectiu de contribuir al desenvolupament de nous fàrmacs mèdics.
El consorci FRAILTECH cerca desenvolupar una tecnologia de detecció precoç de la síndrome de fragilitat basada en arrays d'ADN. Es combinaran tècniques de seqüenciació d'ADN i anàlisi metagenòmics per a crear un conjunt de dades que permeta avaluar el risc de fragilitat. A més, es validarà una calculadora de risc multiescalar que integrarà informació genètica, de microbiota i variables clíniques. El projecte també busca generar bases de dades per a la comunitat d'investigació de fragilitat.
En aquest estudi l'objectiu és el desenvolupament d'una solució ràpida i econòmica per a detectar el risc de deterioració en les extremitats inferiors en persones majors. Utilitzant tecnologies de seqüenciació d'ADN i anàlisi metagenòmic, es crearà un panell de referència que inclourà dades genètiques i microbiòtiques així com una calculadora de risc i una base de dades per a investigacions relacionades.
El projecte LIVERGUT cerca avaluar els efectes del trasplantament de microbioma fecal (FMT) en pacients amb cirrosis. Dos assajos aleatoritzats de fase III seran realitzats per a avaluar la reducció de la hipertensió portal i les complicacions de la cirrosi.
Els organismes multicel·lulars tenen una relació simbiòtica amb microbis, i aquesta associació és essencial per al seu funcionament. Els microbiomes, que són comunitats de microbis, poden classificar-se en tipus específics. Això és rellevant en l'àmbit clínic, ja que ajuda en el diagnòstic, identificació de factors de risc i predicció de l'èxit del tractament. CLOM consisteix en una base de dades de perfils de microbiomes i un classificador de perfils que empra tècniques d'agrupació no supervisades. El principal objectiu és permetre la caracterització i predicció de la microbiota i la seua relació amb malalties, així com per a l'assignació de noves mostres a tipus de comunitats microbianes associades a malalties.
El projecte se centra en l'estudi de les interaccions entre bacteriòfags i bacteris en la microbiota de la panerola Blattella germanica. Es hipotètica que els bacteriòfags afecten la diversitat bacteriana i que la resposta de l'hoste a l’estrés, com els antibiòtics o les temperatures extremes, altera aquestes interaccions i el metabolisme de l'hoste.
Canvis amb l'edat de les interaccions de la microbiota amb el seu hoste humà i determinació d'un nucli permanent de simbionts mutualistes.


 Nudi-Symb
Nudi-Symb
 FRAILTECH
FRAILTECH
 LEGTECH
LEGTECH
 LIVERGUT
LIVERGUT
 CLOM
CLOM
 TEANS-PB
TEANS-PB
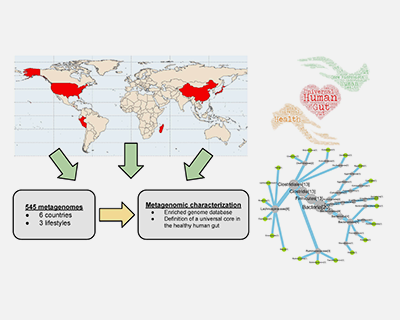 MUTUALCORE
MUTUALCORE