Qué hacemos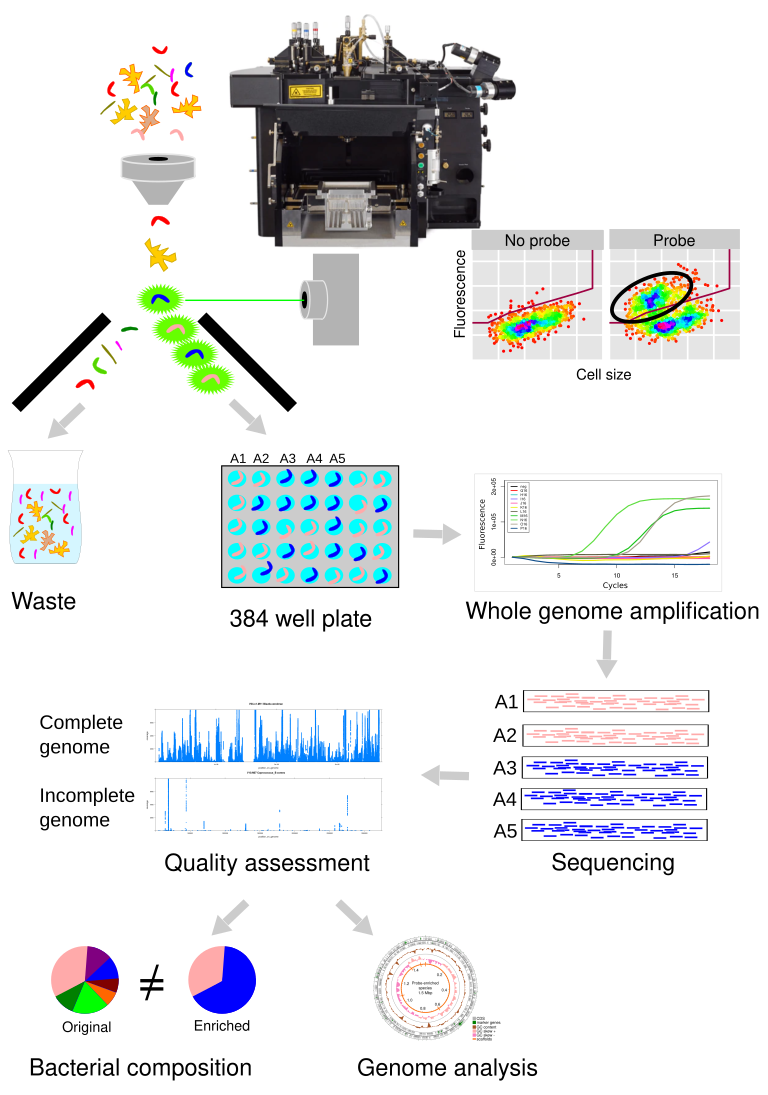
La genómica de células individuales microbianas representa un enfoque innovador para estudiar la diversidad microbiana y la simbiosis entre los microbios. Nos permite obtener genomas de microbios con características de nuestro interés, o detectar relaciones entre microbios que se encuentran uno dentro de otro, o adheridos entre sí.
La genómica unicelular microbiana difiere en gran medida de la genómica unicelular utilizada para el análisis de la expresión génica en las células cancerosas. Los genes expresados en los organismos eucariotas pueden amplificarse mediante cebadores oligo(dT), sin embargo, esto es imposible en el caso de las bacterias, que carecen de las colas poli(A) en sus transcritos.
En el flujo de trabajo de la genómica de células individuales microbianas, se utiliza la clasificación de células activadas por fluorescencia (FACS) para recolectar las células bacterianas de interés, en función de su tamaño celular, su granularidad interna o su fluorescencia, que se analiza por el instrumento FACS a una velocidad de varios miles de células por segundo. Las células se colocan en placas de 96 o 384 pocillos y el ADN se extrae mediante lisis alcalina. Después, se aplica una mezcla de hexámeros aleatorios y phi29 polimerasa en una reacción isotérmica de 4 a 12 horas para enriquecer el ADN por amplificación del genoma completo. Los femtogramos de ADN de una célula se amplifican hasta las cantidades requeridas por los kits de preparación de bibliotecas de secuenciación estándar. El contenido de cada célula individual se secuencia por separado, dando lugar a los denominados genomas individuales amplificados (SAGs). La secuenciación revela la integridad del genoma y su contaminación. Posteriormente, podemos analizar las características metabólicas de los microbios recolectados, o centrarnos en sus interacciones con otros microbios, por ejemplo, los bacteriófagos infectando bacterias, o la presencia de simbiontes adheridos. Sin la genómica de células individuales microbianas no se descubrirían muchos genomas nuevos, ni nuevas interacciones microbianas.
Nuestro grupo domina una amplia variedad de técnicas genómicas. Por ejemplo, a menudo combinamos el flujo de trabajo de la genómica de células individuales microbianas con la metagenómica, que nos proporciona información sobre la composición de la comunidad microbiana y las características genómicas de los genomas ensamblados del metagenoma obtenidos mediante el binning de los contigs (MAGs). Si las muestras contienen muchas células de los hospedadores, como los tejidos de animales o humanos, evaluamos la composición microbiana mediante la secuenciación de amplicones de ADN ribosomal 16S. Además, para verificar nuestras observaciones basadas en la secuencia, a menudo utilizamos técnicas de cultivo tradicionales.
Tenemos colaboradores en todo el mundo, por ejemplo:
- DOE Joint Genome Institute, Lawrence Berkeley National Laboratory, EEUU
- Department of Chemistry and Biochemistry, Universidad de California San Diego, EEUU
- Bigelow Laboratory for Ocean Sciences, EEUU
- Beth Israel Deaconess Medical Center, Facultad de Medicina, Universidad de Harvard, EEUU
- Australian Centre for Ecogenomics, Universidad de Queensland, Australia
- Instituto de biomedicina molecular, Universidad de Commenius, Eslovaquia
Acerca de la responsable del grupo
 Mária Džunková tuvo su primera experiencia con la secuenciación de ADN en la Academia de Ciencias de Chequia durante sus estudios universitarios (2005-2010). Después se trasladó a España para realizar sus estudios de doctorado en la Universidad de Valencia (2010-2016), que incluyeron una estancia de investigación en la Universidad de Harvard (2014). Tras defender su tesis doctoral titulada "Metagenómica del microbioma del intestino humano dirigida por la citometría de flujo", consiguió un postdoctorado en el Australian Centre for Ecogenomics de la Universidad de Queensland (2016-2019), donde desarrolló el método llamado "single-cell viral tagging" que sirve para explorar el rango de hospedador de los bacteriófagos sin necesidad de cultivo microbiano, y lo aplicó al microbioma del intestino humano. Fue invitada a un segundo postdoctorado (2016-2021) en el DOE Joint Genome Institute (Lawrence Berkeley National Laboratory) para continuar con sus técnicas de genómica de células individuales microbianas. En California estudió las relaciones entre los bacteriófagos y las bacterias en muestras ambientales y exploró nuevas técnicas de la genómica de células individuales microbianas para descubrir microbios simbióticos de los animales marinos de cuerpo blando capaces de producir moléculas bioactivas. Se incorporó al Instituto de Biología de Sistemas Integrativos (I2SysBio) en diciembre de 2021 para crear su grupo de investigación de genómica de células individuales microbianas.
Mária Džunková tuvo su primera experiencia con la secuenciación de ADN en la Academia de Ciencias de Chequia durante sus estudios universitarios (2005-2010). Después se trasladó a España para realizar sus estudios de doctorado en la Universidad de Valencia (2010-2016), que incluyeron una estancia de investigación en la Universidad de Harvard (2014). Tras defender su tesis doctoral titulada "Metagenómica del microbioma del intestino humano dirigida por la citometría de flujo", consiguió un postdoctorado en el Australian Centre for Ecogenomics de la Universidad de Queensland (2016-2019), donde desarrolló el método llamado "single-cell viral tagging" que sirve para explorar el rango de hospedador de los bacteriófagos sin necesidad de cultivo microbiano, y lo aplicó al microbioma del intestino humano. Fue invitada a un segundo postdoctorado (2016-2021) en el DOE Joint Genome Institute (Lawrence Berkeley National Laboratory) para continuar con sus técnicas de genómica de células individuales microbianas. En California estudió las relaciones entre los bacteriófagos y las bacterias en muestras ambientales y exploró nuevas técnicas de la genómica de células individuales microbianas para descubrir microbios simbióticos de los animales marinos de cuerpo blando capaces de producir moléculas bioactivas. Se incorporó al Instituto de Biología de Sistemas Integrativos (I2SysBio) en diciembre de 2021 para crear su grupo de investigación de genómica de células individuales microbianas.