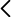BactGem
Título: Modelos metabólicos a escala genómica y ecología bacteriana reversa: aplicación a bacterias de interés económico
Grupos de investigación: Biotecnología y Biología Sintética e Ingeniería Metabólica de Sistemas
Este proyecto contribuirá a la identificación del mecanismo molecular de acción de microorganismos probióticos, basado en la identificación de las vías de síntesis de determinantes moleculares de su acción. Se propone un doble enfoque: computacional y experimental. Así, se elaborará una serie de modelos metabólicos a escala genómica (GEM) a partir de las secuencias anotadas de genomas de bifidobacterias. Esto permitirá obtener las reconstrucciones metabólicas funcionales y analizar, entre otros aspectos, la capacidad de crecimiento, la utilización de fuentes de carbono o los requisitos de factores esenciales. Asimismo, se podrá simular y analizar la producción de metabolitos de interés. Este conocimiento servirá de base para la caracterización de los mecanismos de acción probiótica. De esta manera, será posible obtener mutantes de pérdida de función en los microorganismos de interés, mediante la deleción selectiva de genes candidatos a partir del análisis comparativo de sus genomas y de los modelos metabólicos asociados. Por último, mediante el análisis ecológico reverso, se evaluarán las estrategias de intervención fisiológica. La estrategia general de la ecología reversa también es aplicable a microorganismos que presentan una cinética de crecimiento muy lenta, ya que permite optimizar, primero in silico y luego in vitro, las condiciones de cultivo. Como prueba de concepto, proponemos el diseño de un medio mínimo de cultivo para el fitopatógeno Xylella fastidosa.
Ref.PIE202020E120
Juli Peretó
Patricia Álvarez, Alba Arévalo, Paola Corbín, José Luis García López, Daniel Ramón, Marta Tortajada.
Consejo Superior de Investigaciones Científicas (CSIC).