El refuerzo de la vigilancia genómica del SARS-CoV-2 ha sido respaldado por la Organización Mundial de la Salud (OMS) para incrementar la capacidad de detectar nuevas amenazas y lograr acciones de salud pública de calidad, oportunas y adecuadas.
La detección de virus patógenos para humanos en aguas residuales es una herramienta muy útil y conocida para la vigilancia epidemiológica.
Los objetivos de esta plataforma serán la identificación de mutaciones asociadas a cambios epidemiológicos/clínicos de SARS-CoV-2 con especial atención al fallo vacunal.
Somos parte de un esfuerzo conjunto centrado en comprender mejor los efectos de las mutaciones en el SARS-CoV-2 en tiempo real. Esta beca busca establecer una plataforma para lidiar con amenazas actuales y futuras.
Somos parte de un esfuerzo conjunto centrado en establecer una plataforma para el cribado de antivirales contra amenazas víricas actuales y futuras. El trabajo combina grupos que se encargan del entero proceso de descubrimiento y desarrollo de fármacos.
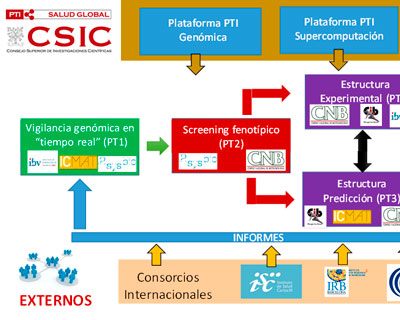
El SARS-CoV-2 es principalmente un virus respiratorio, aunque se ha demostrado que puede replicar en la mucosa intestinal, y ser excretado vía fecal. Datos moleculares han demostrado la presencia de material genético del virus en heces, mientras que los estudios de detección de partículas virales infectivas son aún incipientes y, basados en un número muy pequeño de pacientes.
El grupo de la Dra. Mar Siles Lucas en la IRNASA maneja modelos in vitro e in vivo de interacciones parásito-hospedante con fasciola hepática, que podrían utilizarse para evaluar el potencial del parásito y sus moléculas para modular las rutas de entrada y las rutas de inflamación relevantes en COVID-19. Por una parte, este parásito ha demostrado su influencia en la expresión de las moléculas relacionadas con la endocitosis (por ejemplo, clatrinas) en las células epiteliales de ratones in vitro, rutas que pueden ser pertinentes para la entrada del SARS-Cov-2 en las células humanas. Además, F. hepática da lugar a una respuesta Th2 modificada sin el componente inflamatorio en su huésped in vivo, y esta modulación puede dar lugar a una respuesta inflamatoria controlada a la COVID-19.
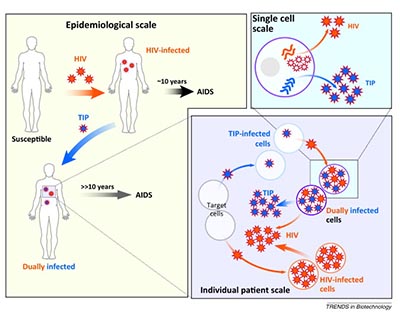
Las partículas de interferencia defectuosas (DIP) son formas degeneradas de genomas virales que no se reproducen pero que siguen siendo infecciosas por la complementación con el virus de tipo salvaje (TW). Las DIP desempeñan un papel importante en la modulación del resultado de la infección y las respuestas inmunitarias, y pueden seleccionarse artificialmente para reforzar su actividad de interferencia y suprimir la replicación del virus completo (partículas de interferencia terapéutica, o TIP por sus siglas en inglés). Proponemos producir DIPs durante la replicación del SARS-CoV-2 en cultivo celular, purificarlas, probar sus efectos antivirales y caracterizarlas molecularmente. El resultado del proyecto será un conjunto específico de TIPs para el SARS-CoV-2 que podría utilizarse inmediatamente para tratar el COVID19.
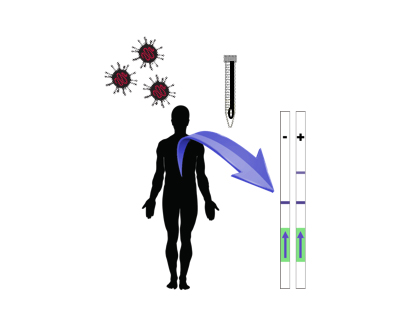
Las técnicas tradicionales de diagnóstico de infecciones virales en la clínica se basan en procedimientos de (RT-)PCR, que llevan mucho tiempo y requieren equipo y recursos humanos muy precisos, lo que impide una intervención rápida y masiva. En este artículo, diseñaremos una nueva clase de biosistemas autónomos destinados a diagnosticar la presencia del SARS-CoV-2. Estos biosistemas consisten en tres pasos de reacción tras la recogida de la muestra sin necesidad de un equipo sofisticado: i) amplificación isotérmica del ARN viral, ii) detección de ácidos nucleicos basada en el CRISPR (que funciona como una reacción de secuenciación sobre la marcha), y iii) revelación de resultados mediante un ensayo inmunocromatográfico.
Aunque el SARS-CoV-2 y otros coronavirus se transmiten fundamentalmente de forma directa entre individuos durante las fases epidémicas, la permanencia del virus en el ambiente tiene el potencial de originar nuevos brotes pese a los esfuerzos de mitigación desplegados. El SARS-CoV-1 se detectó en aguas residuales de hospitales en China y se demostró que las partículas virales podían permanecer infectivas el suficiente tiempo como para constituir un riesgo.
En esta colaboración se aplicará un enfoque multidisciplinario para identificar los inhibidores de la unión y la entrada del receptor del SARS-CoV-2. Combinando los conocimientos de la química médica, la biología química computacional, la biología estructural y la virología, se emplearán tres enfoques paralelos: 1) Ensayo inmediato de las bibliotecas de compuestos (>1.400) mediante un ensayo celular de alto rendimiento para la unión y entrada del receptor del SARS-CoV-2. 2) Examen y diseño, síntesis y evaluación computacionales (3). Desarrollo de inhibidores basados en proteínas derivados del receptor ACE2. La evaluación final de los éxitos en cuanto a eficacia y resistencia a los medicamentos se hará con el SARS-CoV-2.
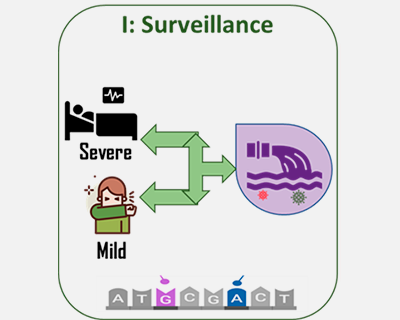
El proyecto científico liderado por investigadores del Consejo Superior de Investigaciones Científicas (CSIC) en colaboración con 40 hospitales de toda España estudiará los genomas comparados del nuevo coronavirus de pacientes con enfermedad Covid19 para entender y predecir la evolución y epidemiología del virus.
El proyecto propone una nueva estrategia para el cribado de fármacos y anticuerpos contra el coronavirus SARS-CoV-2. Se trata de una tecnología innovadora para cribado de fármacos que utiliza un sistema económico, rápido, seguro y eficiente para evaluar todo tipo de compuestos antivirales y anticuerpos que bloquean la entrada del virus SARS-CoV-2 en las células humanas. La utilización de virus recombinantes ha sido empleada con éxito en el cribado de fármacos frente a diferentes virus de importancia médica, así como en el cribado serológico. Las ventajas de la propuesta están bien fundamentadas, destacando que esta tecnología puede ser aplicable en el futuro a otros virus que constituyan un problema de salud pública emergente.


 EvolCovid
EvolCovid
 PTI+: Transmisión y contención
PTI+: Transmisión y contención
 Vigilancia genómica
Vigilancia genómica
 Mutaciones
Mutaciones
 Antiviral platform
Antiviral platform
 FecOrSARS
FecOrSARS
 COVID-19: acción antiinfecciosa y antinflamatoria de las moléculas del parásito inmunomodulador en un formato sintético de uso seguro
COVID-19: acción antiinfecciosa y antinflamatoria de las moléculas del parásito inmunomodulador en un formato sintético de uso seguro
 CoV2TIP
CoV2TIP
 COV-CRISPIS
COV-CRISPIS
 EnvironmentalCOVID
EnvironmentalCOVID
 BlockAce
BlockAce
 SeqCOVID
SeqCOVID
 ANTICOR
ANTICOR