Personal investigador del Instituto de Biología Integrativa de Sistemas (I2SysBio), centro mixto del CSIC y la Universitat de València; del Instituto de Biomedicina de Valencia (IBV) y de la Fundación para el Fomento de la Investigación Sanitaria y Biomédica (FISABIO), entre otros, ha estudiado el efecto que las mutaciones tienen sobre las diferentes cepas de la tuberculosis. Los resultados, publicados en la revista Nature Communications, amplían los conocimientos sobre la evolución de las bacterias causantes de esta enfermedad en humanos.
La tuberculosis es una enfermedad infecciosa causada por la bacteria Mycobacterium tuberculosis, que provoca una mortandad devastadora en humanos y animales, y que también conlleva importantes pérdidas económicas. Conocer cómo se diferencian los distintos linajes bacterianos aumenta la comprensión de los orígenes de la bacteria que causa la enfermedad y los mecanismos genéticos involucrados.
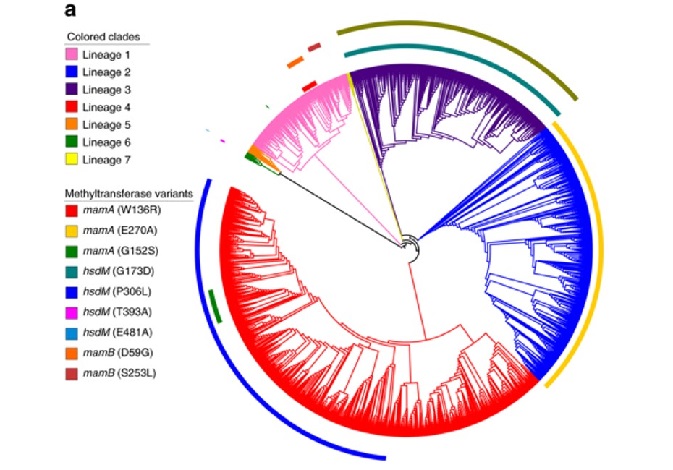
Álvaro Chiner Oms, investigador de la Unidad Mixta Infección y Salud Pública (Instituto de Biología Integrativa de Sistemas, FISABIO-CSISP) y del IBV, destaca que “hemos llevado a cabo una comparación exhaustiva de transcriptomas y ADN-metilomas de diecinueve cepas aisladas representativas del espectro filogenético global de las cepas adaptadas al ser humano del MTBC que nos ha permitido descubrir perfiles transcripcionales únicos”.
El también investigador del CSIC en el IBV Iñaki Comas explica que en las bacterias no recombinantes, donde la mutación suministra la mayor parte de la variación genética, “los procesos selectivos y no selectivos pueden tener un gran impacto en la diversificación funcional. Algunas de estas diferencias funcionales pueden traducirse en características fenotípicas. El complejo Mycobacterium tuberculosis o MTBC, que comprende un grupo de micobacterias que afectan a los humanos y a algunas especies de mamíferos, a pesar de su diversidad extremadamente baja, muestra importantes diferencias biológicas entre cepas y linajes filogenéticos”.
Además, el experto indica: “hemos observado que los patrones de transcripción diferencial entre linajes reflejaban la expresión constitutiva de genes que normalmente están asociados a la virulencia de la bacteria. Aislada de la oportunidad de generar diversidad mediante la transferencia horizontal de genes, la adaptación transcripcional puede permitir que las cepas aisladas de MTBC optimicen su infectividad y transmisión en entornos sutilmente diferentes proporcionados por diferentes poblaciones de huéspedes humanos”. En este sentido, Comas concluye: “nuestro estudio demuestra que la variación en los perfiles transcripcionales de MTBC se debe principalmente a mutaciones en el punto de inicio de la transcripción y probablemente han evolucionado debido a diferencias en las características del huésped”.
Este descubrimiento podría mejorar el conocimiento de cómo las diferentes cepas de la tuberculosis se han adaptado a las poblaciones humanas para transmitirse exitosamente hasta la actualidad.
Artículo:
Álvaro Chiner-Oms, Michael Berney, Christine Boinett, Fernando González-Candelas, Douglas Young, Sebastien Gagneux, William R. Jacobs Jr., Julian Parkhill, Teresa Cortes e Iñaki Comas. «Genome-wide mutational biases fuel transcriptional diversity in the Mycobacterium tuberculosis complex». Nature Communications. DOI: https://doi.org/10.1038/s41467-019-11948-6