
Universitat de València - Grado en Bioqímica y Ciencias Biomédicas
Estructura de Macromoléculas y Enzimología
P1 - Representación y Análisis de Estructuras de Macromoléculas
Protein Data Bank, en www.rcsb.org
Como se ha descrito en la Introducción, la organización internacional wwPDB mantiene un repositorio único de estructuras de macromoléculas obtenidas experimentalmente. Vamos a consultar esa información a través del sitio gestionado por el RCSB (Research Collaboratory for Structural Bioinformatics), cuya base de datos se denomina RCSB PDB.
En esta primera parte conocerás la base de datos y utilizarás alguna de sus herramientas para estudiar estructuras de proteínas.
IMPORTANTE:
Es obligatorio entregar el formulario de resultados individualmente, en pdf, a través de la Tarea correspondiente de Aula Virtual
En la página inicial encontrarás un campo para realizar búsquedas (campo Enter search term(s)) y un menú lateral principal con distintos tabuladores, que también puedes encontrar en el menú de la parte superior.
Los tab→ Welcome y Learn contienen información general. Los tab→ Deposit y Analyze son utilizados por usuarios avanzados. Permiten insertar nuevas estructuras de moléculas, analizar su calidad o comparar distintas estructuras. Entrando en el tab→ Search podemos navegar a través del archivo y localizar estructuras individuales o por tipos. Por último, en el tab→ Visualize encontrarás herramientas sencillas para crear y observar modelos gráficos de estructuras.
Empieza por revisar brevemente la información disponible en la base de datos. Para ello selecciona el tab→ Search (apartado→ PDB statistics) y podrás consultar datos sobre la distribución de las estructuras con arregla a diferentes criterios, como el origen de las estructuras según el organismo de procedencia, los tipos de métodos experimentales que se han utilizado para determinar las estructuras de la base de datos, los tipos de moléculas a los cuales corresponden esas estructuras, etc.
La hemoglobina es una de las proteínas con más estructuras en la base de datos. La primera estructura de hemoglobina fue determinada mediante difracción de rayos X por Max Perutz, galardonado premio Nobel de química (1962) junto a John Kendrew (descubridor de la estructura de la mioglobina mediante el mismo método). Estas estructuras pioneras inauguraron en 1976, junto a otras 10, la primera versión del Protein Data Bank y fueron destacadas como Moleculéculas del Mes en Octubre de 2011.
Quizá te interese saber cuántas otras estructuras de hemoglobina han sido resueltas desde entonces. Vamos a averiguarlo utilizando la herramienta Advanced Search. Entre todas las estructuras que encontremos buscaremos la pionera resuelta por Perutz.
Selecciona el tab→ Search y después → Advanced Search. Comienza buscando todas las estructuras de hemoglobina:
Los registros encontrados aparecen listados en la parte inferior de la página. A la izquierda (o al fondo, dependiendo del tamaño de tu pantalla) verás una columna de controles llamada Refinements, que te permite afinar la búsqueda. Por ejemplo, puedes usar la fecha (RELEASE DATE) para encontrar las primeras estructuras de hemoglobina y localizar entre ellas la de Perutz.
Selecciona el registro de la hemoglobina de Perutz (selecciona el link de su PDB ID). Accederás a una página con información resumida sobre esa estructura, incluyendo datos técnicos sobre su calidad, datos bibliográficos (Literature) y detalles sobre las moléculas que contiene. Entre esto último verás que distingue Macromolecules, es decir, cadenas proteicas, de Small Molecules que se encuentran unidas a las cadenas de proteína (por ejemplo, grupos prostéticos o ligandos de la proteína).
Examina el registro de la hemoglobina de Perutz. Busca información sobre lo siguiente:
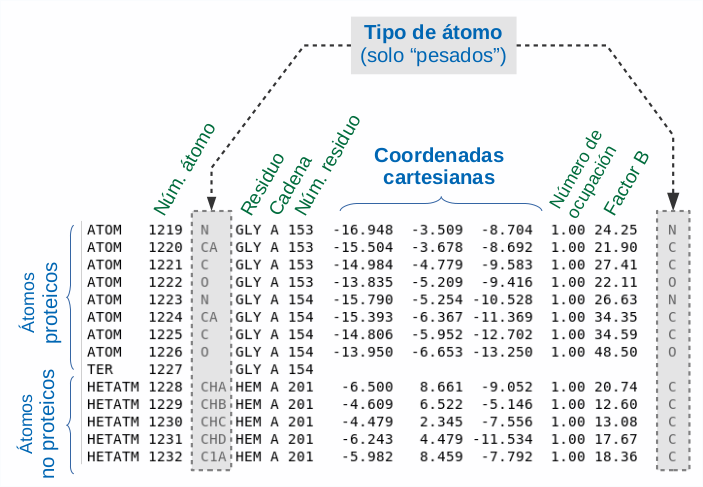
Información estructural en un fichero PDB. Las columnas que contienen las tres coordenadas cartesianas dictan la posición espacial puntual de cada átomo. En las columnas siguientes se encuentran El número de ocupación, que indica la frecuencia de cada posición entre varias conformaciones posibles y el factor B (factor isotrópico de temperatura), que indica el desplazamiento de la posición del átomo con respecto a un valor medio. La información asociada a estos dos últimos números no es necesaria para definir la estructura y se refiere a aspectos relacionados con el desorden y la dinámica de la molécula.
Las estructuras de la base de datos están escritas en ficheros sencillos de texto con formatos especiales. Cuando accedemos a un registro, sus ficheros estructurales se pueden obtener a través del menú desplegable <Display Files>.
El fichero FASTA describe la estructura primaria (secuencia de residuos) de la molécula. Si en la estructura existen varias cadenas poliméricas, cada una de ellas corresponde a una línea continua en el fichero, sin espacios, con excepción de las líneas que comienzan por >, que contienen comentarios.
El fichero PDB describe la estructura tridimensional de la molécula, escrita a través de las coordenadas espaciales o cartesianas {x,y,z} de sus átomos pesados (C, O, N, S, P u otros, si los hay, pero no H). En los ficheros PDB suele haber una primera parte, denominada HEADER con información complementaria (autores, origen de la proteína, método usado, calidad de la estructura, secuencia de residuos, etc). La información estructural se encuentra a continuación y corresponde a todas las líneas que comienzan por ATOM. Si existen grupos químicos no proteicos en la molécula, su estructura se escribe en las lineas que comienzan por HETATM.
Despliega el menú <Display Files> en la página que corresponde a la hemoglobina de Perutz. Selecciona el fichero FASTA Sequence y observa su contenido. Después abre el fichero PDB Format y navega hacia abajo observando su contenido.
Continúa en la Parte 2.
