Universitat de València - Grado en Bioqímica y Ciencias Biomédicas
Estructura de Macromoléculas y Enzimología
P1 -
Representación y Análisis de Estructuras de Macromoléculas
Actualizado:
Parte 2
Representación de modelos moleculares
IMPORTANTE:
- En esta parte tus resultados serán representaciones de la estructura que pueden ser guardadas como "imágenes".
- Guarda tus imágenes al completar cada apartado, ya que vas a necesitar alguna de ellas para completar el formulario de resultados de la práctica que encontrarás en Aula Virtual.
- No es necesario que termines todas las partes en el Aula. Puedes completar o repetir lo que quieras en cualquier otro momento, desde cualquier lugar con acceso a internet.
Es obligatorio entregar el cuestionario de resultados individualmente, en pdf, a través de la Tarea correspondiente de Aula Virtual
Elección de una estructura: Oxi-hemoglobina humana
La hemoglobina de Perutz (2DBH) que acabamos de ver en el apartado anterior fue aislada de sangre de caballo y cristalizada en ausencia de O2. En la base de datos existen también estructuras de hemoglobina humana con O2 como ligando (oxi-hemoglobina). Para trabajar el resto de la práctica vamos a buscar una de ellas, que además haya sido determinada con un alto nivel de resolución (al menos 1.3 Å).
Selecciona de nuevo el tab→ Search y después → Advanced Search y procede de la manera que sigue:
- En el Menú desplegable→ selecciona Structure Details→ Structure Title y escribe et término human hemoglobin.
- Añade ahora un nuevo campo de búsqueda (Add Attribute) y selecciona en el Menú desplegable→ Methods→ Refinement Resolution con la condición < 1.3
- Entre la lista de estructuras que consigues, elige una que contenga O2 unido (oxy form)
La estructura que has elegido se puede representer leyendo la información del fichero PDB con ayuda de un programa informático adecuado. La base de datos RCSB proporciona herramientas de representación y visualización in situ optimizadas para una observación y análisis sencillos de estructuras de macromoléculas. Accede a las herramientas de visualización a través del tab→ Structure. Observarás una representación estandarizada de tu estructura en la ventana de visualización de <Mol*> (Mol-star).
Podrás generar tus propias representaciones de la molécula, resaltar partes de interés, observar todo ello a distintas escalas desde cualquier ángulo de rotación y convertir tus representaciones en imágenes ilustrativas.
➀
Manejo básico del programa de representación de estructuras
Accede ar <Mol*> a través del tab→ Structure
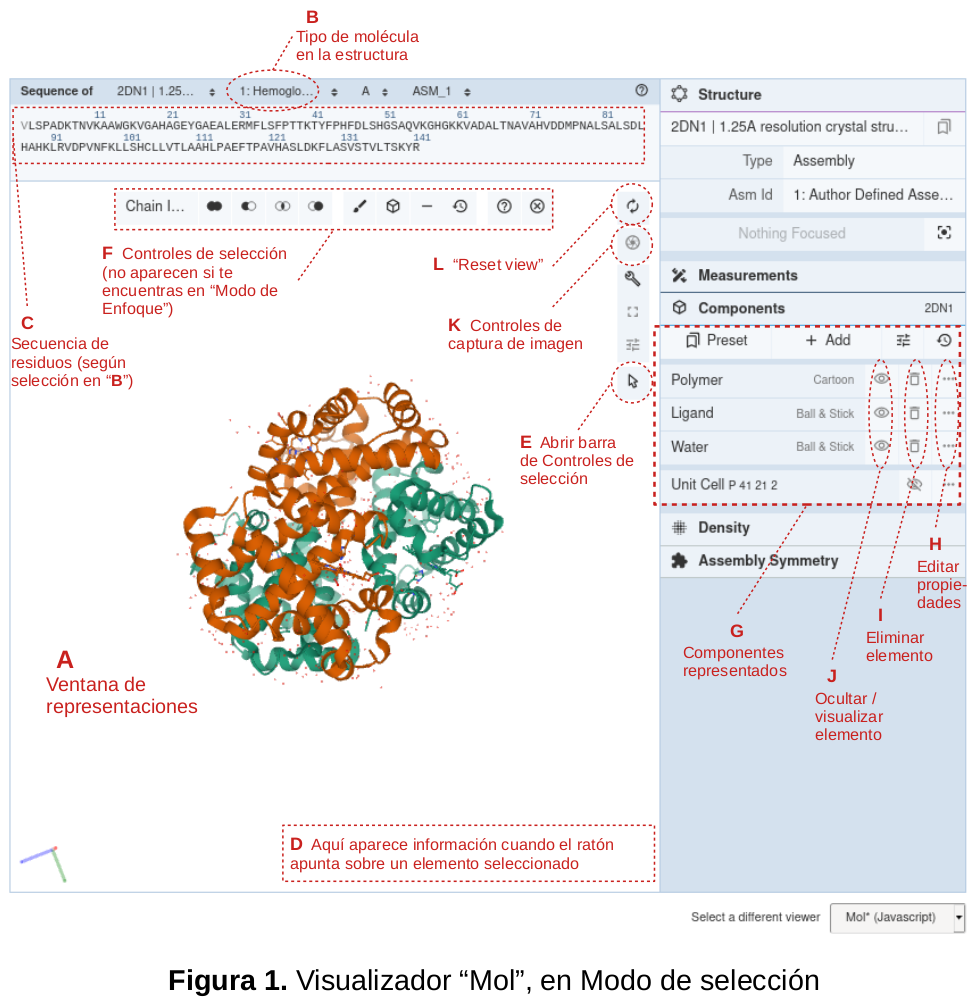
Observarás una ventana que contiene una representación por defecto de la estructura (A en la Figura 1). Prueba a rotarla, reescalarda o moverla con los controles básicos de tu ratón (ver cuadro guía). Desde la parte superior (B en la Figura 1) puedes elegir los distintos "tipos de molécula" que contine la estructura.
Si se trata de una cadena de proteína, se mostrará su secuencia de aminoácidos (C), en la que cada letra es un residuo de aminoácido. Si posicionas el cursor sobre uno de esos residuos lo verás resaltado en la secuencia y en la representación y se mostrará información sobre él (D). Dependiendo del Modo de respuesta (ver cuadro guía), haciendo click sobre un elemento la representación te muestra una vista ampliada con detalles de sus interacciones (enfoque) o bien el elemento quedará seleccionado.
Guía-1. Uso Básico
Funciones del ratón
Con el puntero en la ventana de representaciones (A, en la Figura 1)...
- Rotar la molécula: con botón izquierdo→ click + mover
- Reescalar la molécula: con botón central → girar la rueda
- Desplazar la molécula: con botón derecho → click + mover
- Señalar. Mueve el cursor sobre cualquier elemento (de la secuencia o la imagen). Se mostrará información sobre ese elemento
en la zona D de la ventana de representaciones.
Modo de Enfoque y Modo de Selección
Cuando haces click sobre un elemento hay dos tipos de respuesta según te encuentres en Modo de Selección o en Modo de Enfoque. Para cambiar de modo haz click.→ sobre la flecha blanca de la barra vertical de controles (E en la Figura 1):
- En Modo de Enfoque (por defecto), si haces click en un elemento (residuo de la secuencia o sobre la imagen, o bien uno de los Componentes que aparecen en G), la representación se centra en ese elemento. Si se trata de una parte (átomo o residuo) se mostrarán detalles a su alrededor.
- El Modo de Selección se activa por la flecha blanca de la barra vertical E, que hace visible la barra horizontal F. En este modo, si haces click sobre un elemento (en la secuencia o sobre la imagen), dicho elemento quedará seleccionado. La selección depende de los valores que elijas con los controles de selección (F). Con los elementos que selecciones puedes crear nuevos componentes representados (G). Todos los componentes representados pueden ser editados (H), eliminados (I) u ocultados (J).
Intercambia entre los modos de Selección y Enfoque a través de la flecha blanca de la barra vertical de controles (E), o sal del modo Selección con el control ⊗ de la barra horizontal F.
Captura de imágenes
Para guardar una imagen de una representación, utiliza el control K, que te permitirá copiar la imagen o bien guardarla en un fichero. Utiliza esta función para guardar tus representaciones cuando creas que son suficientemente claras.
Reset: Retorno a condiciones iniciales
Cuando algo vaya mal y quieras recuperar tu estado inicial...
- Si quieres recuperar la visión estándar en la ventana de representaciones, haz click en L.
- Si deseas volver al estado inicial de tu representación, con los componentes originales, en G, elige la opción Preset→ Automatic
Ahora que conoces un poco el programa vamos a representar la estructura de la hemoglobina, paso a paso, desde el nivel primario al cuaternario.
➁
Recrear la cadena, desde el primer residuo
Empezaremos representando los primeros residuos desde el extremo N-terminal, prestando atención a los detalles de la estructura (enlace peptídico, indicios de estructura secundaria).
Primero es conveniente eliminar o esconder la representación que aparece por defecto. Además, para poder seleccionar de manera individual las cadenas alfa (α) y beta (β) (denominadas A y B), nos aseguramos de que en la parte superior se encuentra seleccionada solo una de ellas, la que corresponde a la unidad de asimetría 1 (ASM_1).
Guía-2.1. Primeros residuos
- En el marco superior, derecha, debe aparecer Sequence of - 2DN1 | Chain | Hemoglobin alpha | A | ASM_1
(B en la Figura 1). .
- Esconde todos los Componentes representados (J en la Figura 1 de la Guía 1). También puedes eliminarlos (pulsando sobre I, o bien a través de Components → Preset → Empty.
- Asegúrate de estar en Modo de selección (ver Guía 1).
- Elige Residue como nivel de selección (primer campo en el cuadro de selección, F en la Figura 1)
- En la secuencia (C en la Figura 1), selecciona el primer residuo.
NOTA: Aunque la primera letra en la secuencia es V (Val1), verás que aparece con color gris pálido. Se debe a que ese primer residuo no está presente en el fichero de coordenadas (pdb). El primer residuo que sí aparece y sí puedes representar es L (Leu2). La explicación a esta aparente contradicción está en el REMARK 465 del fichero pdb, que puedes leer a través del menú desplegable <Display Files>.
- Crea un nuevo componente con tu selección:
Components → Add
⇒ Representation → click en Create Later y selecciona Ball & Stick
⇒ Options → Label
→ Escribe el nombre que quieras, por ejemplo Leu2
⇒ click en Create Component
Verás el residuo Leu2 en la ventana de representaciones.
- Observa tu representación. Mueve el cursor sobre el residuo. Rotalo o cambia su escala. Para saber cuáles son sus átomos, cambia a Atom como nivel de selección (primer campo en el cuadro de selección, F en la Figura 1). Después pasa el cursor sobre los átomos de la representación.
Guía-2.2. Enlace peptídico
- Añade mas residuos a tu representación:
Elige de nuevo Residue como nivel de selección, como en el punto (4).
Selecciona los residuos tercero y cuarto (Ser y Pro), como en el punto (5)).
Añade esta nueva selección a tu componente anterior. Para ello, edita las propiedades del componente (H en la Figura 1), selecciona Modify by Selection y después → Include
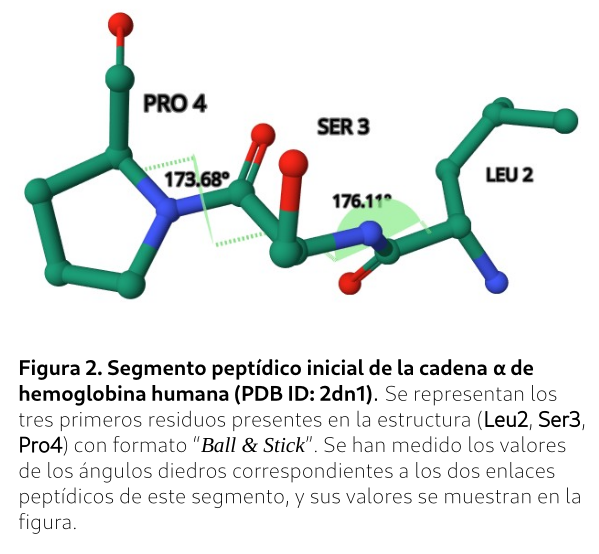
- En este fragmento de 3 residuos hay dos enlaces peptídidos. Vamos a medir la torsión (ángulo diedro) de cada uno de ellos:
Elige Atom como nivel de selección.
En la estructura, click en Cα y C-carbonilo de L2 y en N-amida y Cα de S3.
Selecciona Measurements (menú principal de la derecha) y después Add→ Dihedral. Verás que en la representación aparece marcado el ángulo diedro que corresponde al primer plano peptídico.
Selecciona los 4 átomos que corresponden al segundo enlace peptídico y añade el correspondiente ángulo diedro.
Guarda tu Resultado
Cuando hayas completado esta parte...
- Busca una orientación clara de tu estructura en la ventana de representaciones y exporta la imagen. Para ello, sigue las instrucciones sobre captura de imágenes que se encuentran al final de la Guía-1
- Ten en cuenta que si el formulario de resultados te pide esta imagen deberás incluir también un texto que la describa (tienes un ejemplo en el pie de la Figura 2
➂
Representación de Estructuras Secundaria y Terciaria
Las representación anterior contiene solo unos pocos residuos. Si alargamos la cadena con algunos residuos mas podremos observar propiedades de estructura secundaria. Para estudiar este tipo de estructura utilizaremos una representación de tipo cartoon y prestaremos atención a la existencia de enlaces de hidrógeno característicos.
Por otro lado, si continuamos alargando la cadena hasta el final tendríamos una disposición espacial que corresponde a la estructura terciaria. Para estos niveles superiores de estructura son necesarios otros tipos de representación más adecuados.
Guía-3. Estructura Secundaria
- Alarga tu representación con tres residuos mas: En la secuencia selecciona ADK (hasta el residuos Lys7. Depués, para el Componente con el que has trabajado antes, selecciona:
⇒ Modify Selection → Include
Como puedes ver la cadena continua con un giro. Veamos qué estructura presenta.
- Añade una representación de estructura secundaria (Cartoon): Elige el Componente anterior.
Edita sus propiedades (click en H, ver Figura 1). En el menú que aparece...
⇒ Add Representation → Cartoon.
Verás que en la ventana de representaciones aparece una cinta que a partir del residuo de Ser presenta un giro helicoidal. El color de esa representación corresponde por defecto al tipo de cadena. Si queremos que el color informe sobre el tipo de estructura secundaria debemos asignarlo de manera adecuada.
- Colorea por tipo de estructura secundaria: Accede de nuevo a las propiedades del Componente: click en H...
⇒ Set Coloring →
Verás seleccionado Chain Property → Chain Id
Cambia la selección a Residue Property → Secondary Structure
Para saber el significado de cada color:
⇒ click en H (propiedades del componente). En la lista de propiedades...
⇒ Cartoon Representation → Color Theme ⇒ click en "..." (propiedades del tema de color). Cambia Default a Custom y despliega "..." (propiedades de los colores).
Verás la asignación de colores por defectosegún la estructura secundaria. Por ejemplo:
magenta → hélice α (hélice 3,613)
morado → hélice 310
blanco → coil
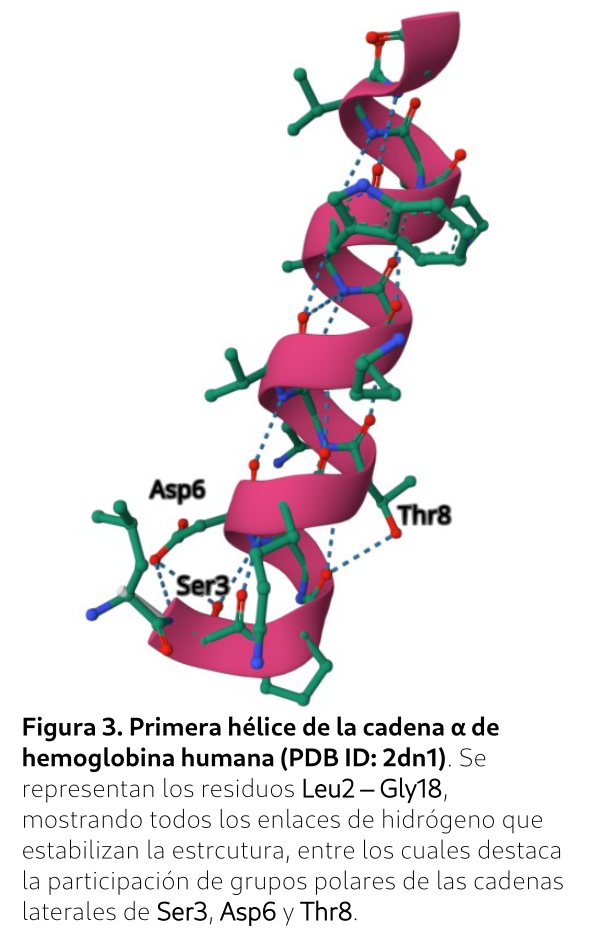
- Enlaces de hidrógeno intra-hélice: Recuerda que una hélice α debería haber enlaces de H entre residuos i e i+4. Veamos si es así:
⇒ Add Representation → Non-covalent Interactions.
NOTA: Observa dos cosas interesantes:
(i) En este fragmento hay un solo enlace de H entre grupos C=O y N-H peptídicos (de tipo i -- i+4).
(ii) Sin embargo, hay varios enlaces de H en los que participan grupos polares de las cadenas laterales. Estos últimos tipos de enlace aparecen con frecuencia estabilizando los extremos de las hélices α.
- Alarga aún mas la cadena. Continua añadiendo residuos a tu Componente hasta observar la primera hélice α completa y las hálices que siguen a esta, con los loops entre hélices.
NOTA: Observarás que la tercera hélice es mas corta y distinta de las demás. Se trata de un segmento de hélice 310 que se diferencia del resto de hélices de la cadena, de tipo hélice-α (3.613). Compara los enlaces de H en ambos tipos de hélice.
Guarda tu Resultado
Cuando hayas completado esta parte busca una orientación clara de tu estructura en la ventana de representaciones y exporta la imagen. Acuérdate de que si el formulario de resultados te pide esta imagen deberás incluir también un texto que la describa (tienes un ejemplo en el pie de la Figura 3
➃
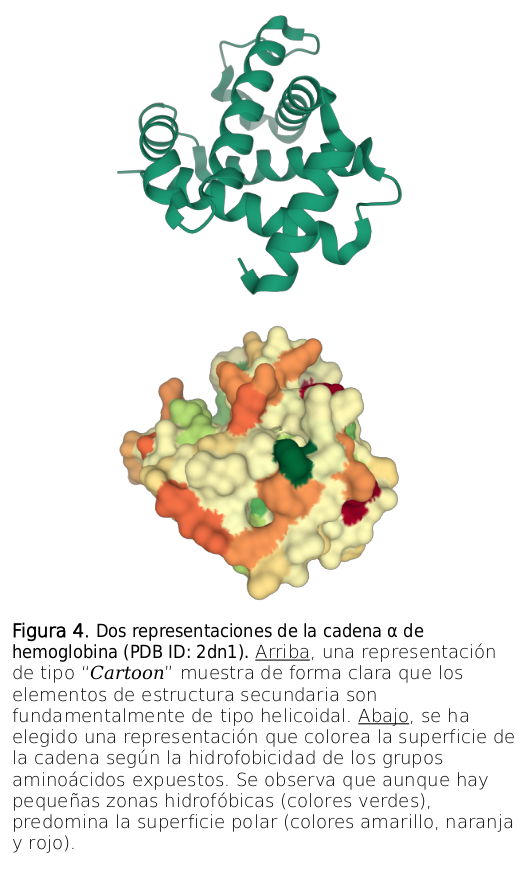
Estructura Terciaria
Si alargas la cadena hasta el final, completando el punto (5) de la Guía-3), tendrás una representación de una cadena polipeptídica completa, aunque bastante esquemática. Por ello vamos a visualizar la superficie de la molécula con propiedades de los residuos expuestos:
Guía-4. Estructura Terciaria
- Asegúrate de que tu representación anterior contiene una cadena completa. Si no es así, complétala.
- Añade una representación de superficie: Elige el Componente que contiene tu cadena completa.
Edita las propiedades de ese componente (click en H, ver Figura 1). En el menú que aparece...
⇒ Add Representation → Molecular Surface.
Verás que aparece la superficie externa la cadena con un color homogéneo.
- Colorea la superficie molecular. Para ello, de nuevo
Edita las propiedades del Componente que contiene tu cadena completa (como en el punto 2). Al fondo del menú desplegable que aparece, selecciona...
⇒ Molecular Surface Representation y a la derecha de Color Theme cambia Chain Id por Residue Propertie → Hydrophobicity.
NOTA-1: En la representación por Hydrophobicity que usa el programa se representa la polaridad con una escala de colores que va desde lo mas hidrofóbico, en verde a lo mas hidrofílico (polar), en rojo .
NOTA-2: Si en vez de asignar el color usando el método del punto 3), lo haces a través de Set Coloring (como en el punto (2) de la Guía-3), la nueva coloración afectará a todas las representaciones del componente.
Rota la estructura y observa la presencia de cavidades y la distribución de polaridad en la superficie. En concreto, fíjate en la hidrofobicidad de la cavidad que corresponde al grup hemo.
Observa también que en la superficie de la cadena no solo hay residuos polares, debido en parte a que las zonas hidrofóbicas quedan cubiertas en la estructura cuaternaria.
Guarda tu Resultado
Cuando hayas completado esta parte busca una orientación clara de tu estructura en la ventana de representaciones y exporta la imagen. Acuérdate de que si el formulario de resultados te pide esta imagen deberás incluir también un texto que la describa (tienes un ejemplo en el pie de la Figura 4
➄
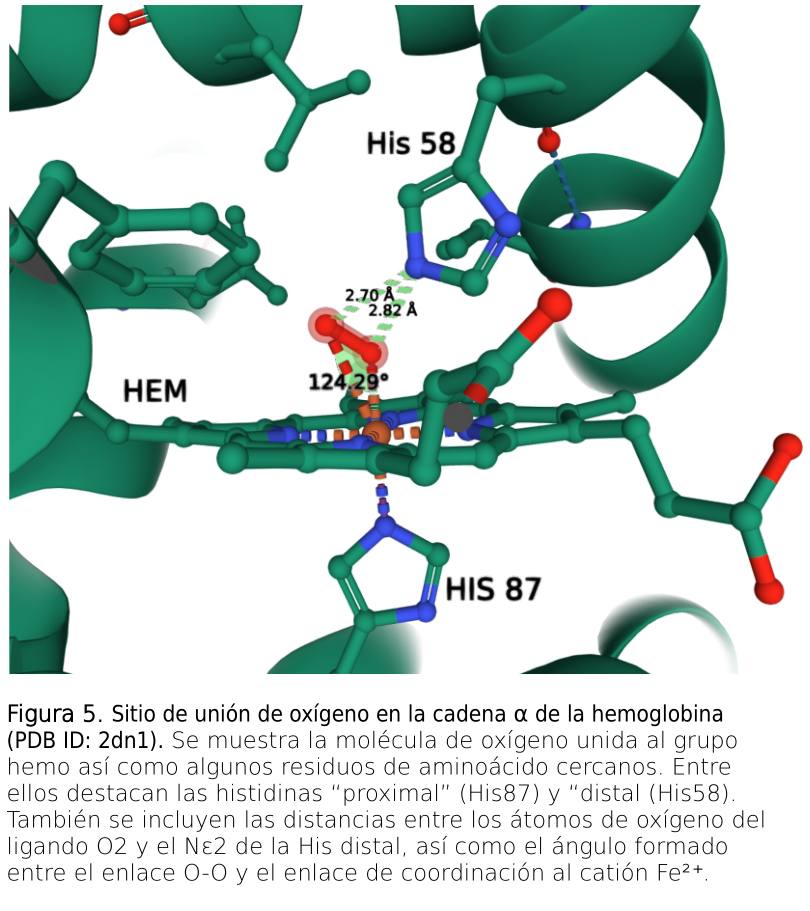
Sitio de Unión de Oxígeno en la Hemoglonina
En la oxi-hemoglobina la molécula de O2 se encuentra unida mediante enlace de coordinación al catión Fe2+ del grupo hemo. Vamos a representar con detalle el sitio de unión de O2 en cualquiera de las dos cadenas, α o β.
Guía-5. O2 unido al grupo hemo
- Esconde todos los Componentes representados (J en la Figura 1 de la Guía 1). También puedes eliminarlos (pulsando sobre I, o bien a través de Components → Preset → Empty.
- En la parte superior-derecha, haz click junto a Type y selecciona Model. En la ventana aparece el dímero α-β.
- Oculta el Componente Water. Deja visible Polymer y Ligand.
- En la parte izquierda de la barra de selecciones (F, Figura 1), elige → Residue.
- Activa el Modo de Enfoque (click en botón E, Guía-1 y Figura 1).
- Localiza la molécula de oxígeno en cualquiera de las dos cadenas (ver Figura 5). Haz un clic sobre ella con el botón izquierto del ratón.
Verás como la ventana de representaciones enfoca la zona donde se encuentra la molécula de oxígeno, mostrando todas las partes cercanas. Puedes rotar o reescalar la estructura hasta verlo claro.
- Marca Residuos o Átomos. Entra en Modo Selección y elige Atom o Residue (dependiendo de lo que quieras marcar). Haz click sobre aquello que quieras marcar y selecciona Measurements (menú principal de la derecha) y después Add→ Label
NOTA: Deberías ver tu nueva etiqueta en la ventana de representaciones, junto a aquello que querías marcar. Si no ves nada prueba a girar tu representación hasta que lo veas. El formato de la etiqueta se puede modificar seleccionando las opciones de edición (símbolo ... de las opciones de tu nueva etiqueta).
Utilizando esta estrategia, introduce etiquetas para las Histidinas distal y proximal. La His proximal se encuentra unida por enlace de coordinación al Fe2+ del grupo Hemo. La His distal se encuentra en una posición opuesta a la anterior, cercana a la molécula de oxígeno.
- Mide y marca Distancias. Puedes introducir una etiqueta de distancia entre dos átomos através de
⇒ Measurements→ Add→ Distance (similar a lo indicado en el apartado anterior). Sin embargo, debes seleccionar primero dos átomos: Elige Atom y la opción Add/Union selection en la barra horizontal F.
Utilizando esta estrategia, mide / marca las distancias entre cada átomo de la molécula de O2 y el Nε2 de la His distal.
- Mide y marca Ángulos. Para introducir una etiqueta de ángulo, procede con
⇒ Measurements→ Add→ Angle, habiendo seleccionado primero tres átomos (Ver punto (6)).
Utilizando esta estrategia, mide / marca el ángulo que forman el enlace O—O de la molécula de O2 y el enlace de coordinación de la molécula de O2 con el catión Fe2+ del grupo Hemo. Las Etiquetas de distancias y ángulos se mostrarán en tu representación.
Guarda tu Resultado
Cuando hayas completado esta parte busca una orientación clara de tu estructura en la ventana de representaciones y exporta la imagen. Acuérdate de que si el formulario de resultados te pide esta imagen deberás incluir también un texto que la describa (tienes un ejemplo en el pie de la Figura 5