
Universitat de València - Grado en Química
Prácticas de Bioquímica
P1 - Representación y Análisis de Estructuras de Macromoléculas
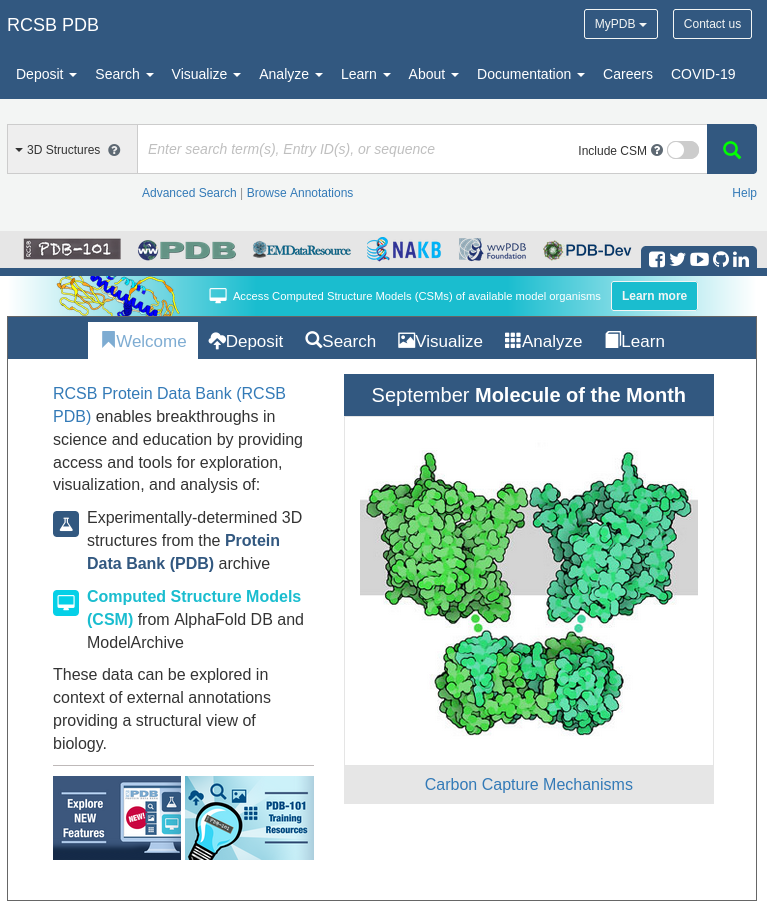
Protein Data Bank, en www.rcsb.org
Como se ha descrito en la Introducción, la organización internacional wwPDB mantiene un repositorio único de estructuras de macromoléculas obtenidas experimentalmente. Vamos a consultar esa información a través del sitio gestionado por el RCSB (Research Collaboratory for Structural Bioinformatics), cuya base de datos se denomina RCSB PDB.
En esta primera parte conocerás la base de datos y utilizarás alguna de sus herramientas para estudiar estructuras de proteínas.
IMPORTANTE:
Es obligatorio entregar el formulario de resultados individualmente, en pdf, a través de la Tarea correspondiente de Aula Virtual
En la página inicial encontrarás un campo para realizar búsquedas (campo Enter search term(s)) y un menú principal con distintos tabuladores, que también puedes encontrar en la parte superior.
Los tab→ Welcome y Learn contienen información general. Los tab→ Deposit y Analyze son utilizados por usuarios avanzados. Permiten insertar nuevas estructuras de moléculas, analizar su calidad o comparar distintas estructuras. Entrando en el tab→ Search podemos navegar a través del archivo y localizar estructuras individuales o por tipos. Por último, en el tab→ Visualize encontrarás herramientas sencillas para crear y observar modelos gráficos de estructuras.
Empieza por revisar brevemente la información disponible en la base de datos. Para ello selecciona el tab→ Search (apartado→ PDB Statistics) y averigua:
Cada mes la base de datos selecciona una molécula "estrella" (Molecule of the Month), la cual se accede facilmente desde la página inicial de RCSB-PDB. Utilizaremos la molécula destacada este mes como primer ejemplo de estructuras interesantes de proteínas. Accederás a un breve artículo sobre esa molécula que explica cómo es y qué función tiene a través de sus estructuras presentes en la base de datos. Cuando el texto de ese artículo comenta sobre una estructura, menciona también su identificador en la base de datos (PDB ID), al cual asocia un link que te permite llegar con un solo click al registro de la estructura, donde podrás encontrar todos los detalles sobre ella.
NOTA: Todas las Moléculas de meses anteriores se encontran através del tab→ Learn (link→ Molecule of the Month)
Selecciona el link de la molécula de este mes (selecciona la imagen de Molecule of the Month). Lee la página sobre esta molécula y responde:
Vamos a trabajar el resto de la práctica con la estructura de una proteína que seguramente ya conoces: la hemoglobina. Un pequeño problema es que la base de datos contiene muchas estructuras de esta molécula. Buscaremos una de origen humano, que contenga oxígeno unido (oxi-hemoglobina) y que haya sido determinada con una alto nivel de resolución (al menos 1.3 Å). Para ello vamos a utilizar la herramienta de búsqueda avanzada.
Selecciona el tab→ Search y después → Advanced Search.
Comenzaremos buscando human hemoglobin. Para ello, en el apartado Structure Attributes abre el Menú desplegable y selecciona Structure Details→ Structure Title.
Verás que el número de estructuras que encuentra es muy grande. Todas ellas corresponden a hemoglobina humana, pero han sido investigadas con distintos ligandos, en disintas condiciones..., etc.
Añade ahora un nuevo campo de búsqueda (Add Attribute) y en su Menú desplegable selecciona Methods→ Refinement Resolution, añadiendo la condición < 1.3:
Entra en el registro de la estructura de la oxi-hemoglobina humana que has encontrado en el apartado anterior (selecciona el link de su PDB ID). Al acceder al registro encontrarás información resumida sobre esa estructura, incluyendo información técnica sobre su calidad, información bibliográfica (Literature) y detalles sobre las moléculas que contiene. Entre esto último verás que distingue entre Macromolecules (cadenas proteicas) y Small Molecules. Estas últimas son las que se encuentran unidas a las cadenas de proteína (es decir, ligandos de la proteína).
Examina el registro de la oxi-hemoglobina humana que has encontrado . Busca información para responder a las siguientes preguntas:
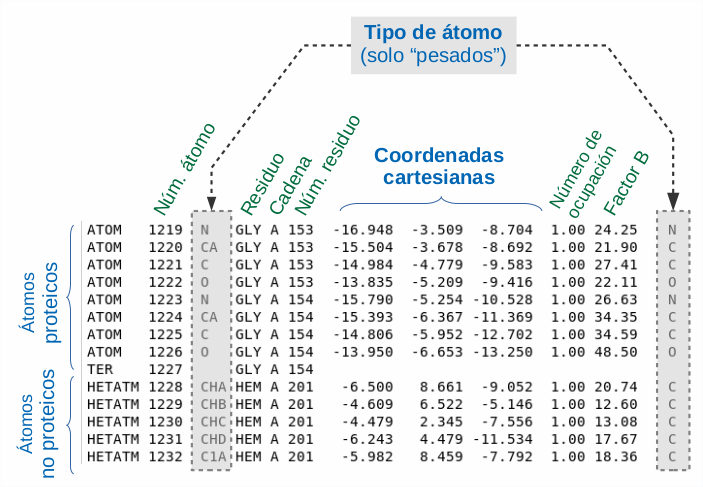
Información estructural en el formato PDB. Las columnas que contienen las tres coordenadas cartesianas dictan la posición espacial puntual de cada átomo. El número de ocupación se refiere a la frecuencia de cada posición espacial entre varias conformaciones posibles en la estructura y el factor B (factor isotrópico de temperatura) indica el desplazamiento de la posición del átomo con respecto a un valor medio.
Las estructuras de la base de datos están escritas en ficheros sencillos de texto con formatos especiales. Cuando accedemos a un registro, sus ficheros estructurales se pueden obtener a través del menú desplegable <Display Files>.
Entre los ficheros estructurales de cada registro los mas útiles y sencillos de entender corresponden a los formatos FASTA y PDB.
El fichero FASTA describe la se encuentra estructura primaria (secuencia de residuos) de la molécula. Si en la estructura existen varias cadenas poliméricas, cada una de ellas corresponde a una línea continua en el fichero, sin espacios, con excepción de las líneas que comienzan por >, que contienen comentarios.
El fichero PDB describe la estructura tridimensional de la molécula escrita a través de las coordenadas espaciales o cartesianas {x,y,z} de sus átomos pesados (C, O, N, S, P u otros, si los hay, pero no H). En los ficheros PDB suele haber una primera parte, denominada HEADER con información complementaria (autores, origen de la proteína, método usado, calidad de la estructura, secuencia de residuos, etc). La información estructural se encuentra a continuación y corresponde a todas las líneas que comienzan por ATOM. Si existen grupos químicos no proteicos en la molécula, su estructura se escribe en las lineas que comienzan por HETATM. En los casos en los que aparece información en líneas marcadas con ANISOU, esta corresponde a los factores de temperatura anisotrópicos de cada átomo, (desplazamiento de la posición del átomo con respecto a su posición media) que proporcionan información acerca de la flexibilidad de la molécula.
Despliega el menú <Display Files> del registro que acabas de encontrar. Selecciona el fichero FASTA Sequence y observa su contenido. Después abre el fichero PDB Format y navega hacia abajo observando su contenido.
Manteń abierta la página del registro estructural y continúa en la Parte 2.
