
Universitat de València - Máster en Aproximaciones Moleculares en Ciencias de la Salud
Tecnologías de la Medicina Molecular
Modelización y Análisis de Estructuras Macromoleculares
Todas las estructuras de macromoléculas obtenidas experimentalmente se encuentran depositadas en un único archivo y pueden accederse de manera gratuita para su utilización. La cantidad de información que ello supone es enorme, pero es mucho mayor aún la información que puede obtenerse a través de su análisis detallado. Dicho análisis estructural requiere la creación de modelos moleculares a escala, lo cual se lleva a cabo con la ayuda de programas informáticos.
Puedes encontrar mas detalles sobre la información estructural en este enlace.
En esta sesión aprenderemos de manera básica, a través de un ejemplo interesante,
(i) cómo conseguir información estructural, con alta resolución, de una proteína.
(ii) en qué consiste esa información
(iii) cómo podemos aprovecharla para investigar / entender esa proteína.
El archivo único de estructuras de macromoléculas Biológicas es gestionado por el WorldWide Protein Data Bank (wwPDB). Accederemos a él a través de RCSB PDB. En este sitio web, además de realizar búsquedas de estructuras concretas, tenemos disponible de manera directa información general sobre las estructuras y podemos llevar a cabo su visualización de manera sencilla.
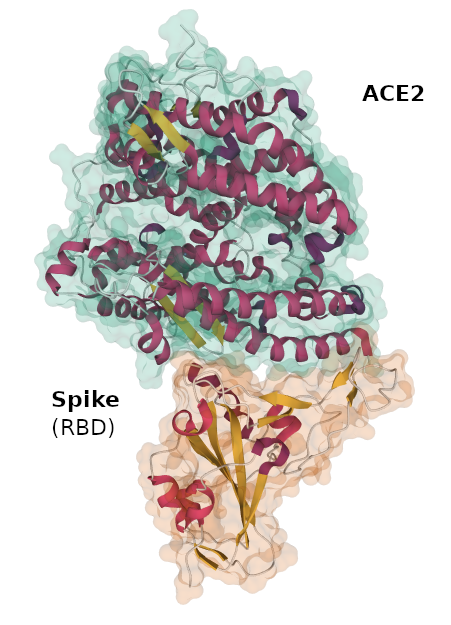
Vamos a tomar como ejemplo de trabajo durante toda la sesión la estructura de un complejo proteico de gran interés, formado por la glicoproteína spike del coronavirus SARS-Cov-2 (proteína S) y el dominio receptor (extramembrana) de la enzima-2 convertidora de angiotensina humana (hACE2). La proteína S es la principal responsable de la entrada del virus en células humanas. Para ello reconoce específicamente a hACE2 (abundante en células pulmonares, de corazón y riñón) que actúa como receptora del vírus. La unión de ambas facilita el canbio conformacional necesario para la fusión entre las membranas del vírus y de la célula hospedadora.
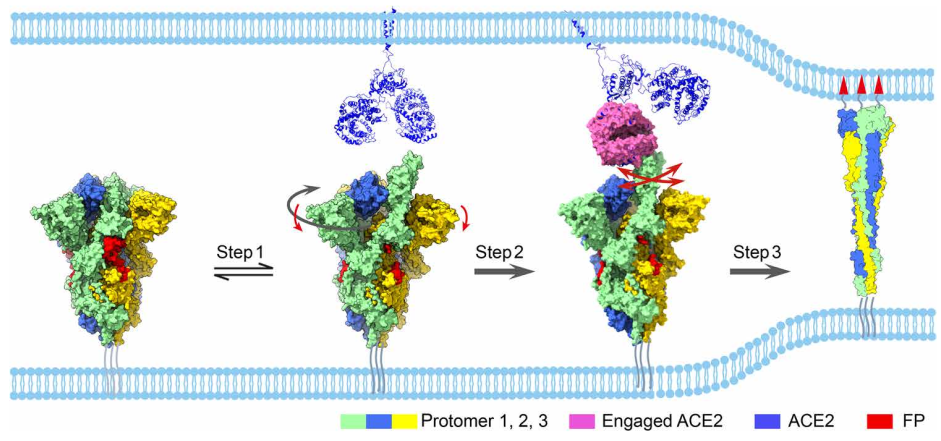
Inducción de la transición conformacional de la protéína-S a través de su unión a ACE2 (Xu et al., Sci. Adv. 2021; 7 : eabe5575).
Este complejo es probablemente el evento más importante para la elevada infectividad del virus. Por ello, desde el inicio de la pandemia de Covid-19 se está investigando con gran intensidad.
Existen centenares de estructuras del complejo publicadas desde Marzo de 2020, resueltas mediante difracción de rayos-X o mediante criomicroscopía electrónica. Algunas de estas estructuras contienen el trímero de la proteína S. En otras se ha utilizado solo el dominio de unión al receptor (RBD). Además, existen también estructuras de distintas versiones de hACE2, así como de la proteína S en estados cerrado (prefusión) o abierto, tanto del SARS-CoV-2 como del virus SARS-CoV.
El complejo fue "nombrado" Molécula del Mes en junio de 2020. Puedes aprovechar la lectura del artículo elaborado con tal motivo para informarte sobre su contexto biológico. También puedes encontrar más información en un artículo reciente sobre las propiedades dinámicas de esta estructura.
Continuar... Parte 1. Acceso a las Estructuras
