
Universitat de València - Máster en Aproximaciones Moleculares en Ciencias de la Salud
Tecnologías de la Medicina Molecular
Modelización y Análisis de Estructuras Macromoleculares
Como acabas de ver en la Parte 1, aunque es posible representar la estructura a través de herramientas presentes en la propia base de datos, sus posibilidades son muy limitadas. Por ello vamos a utilizar el programa VMD (Visual Molecular Dynamics), desarrollado en la Universidad de Illinois y disponible en código abierto de forma libre. Puedes encontrar un manual de VMD en este enlace.
Para continuar necesitarás utilizar un ordenador con sistema operativo Windows, MacOSX o Linux. Descarga e instala en tu propio ordenador la versión 1.9.3 de VMD que encontrarás en este enlace. Elige la descarga adecuada para tu equipo (preferiblemente una opción sin CUDA). El programa se ofrece de manera gratuita, aunque necesitarás registrarte y aceptar los términos de la licencia.
Con ayuda de VMD representaremos y analizaremos la estructura de la oxi-hemoglobina (pdb-ID 2DN1). Dado que las coordenadas disponibles en la base de datos corresponden a una unidad de asimetría dimérica, mientras que el oligómero funcional de la hemoglobina es un tetrámero (Ver apartado sobre Estructura Cuaternaria en La Información Estructural), hemos calculado a parte la estructura tetramérica. Puedes descargar el fichero de coordenadas de dicha estructura a través de este enlace.
Al abrir con VMD el fichero PDB del tetrámero de oxi-hemoglobina obtendrás inicialmente una representación por defecto de todos los atomos de la molecula. Vamos a crear representaciones más claras teniendo en cuenta aquello que nos interese observar o analizar. Para ello utilizaremos las herramientas que aparecen en la ventana de Graphics -->Representations. Podemos combinar múltiples tipos de representación, de toda la molécula o de partes de ella utilizando adecuadamente las reglas de selección para cadenas, residuos, etc.
Para empezar a familiarizarte con todo ello sigue, paso a paso, las instrucciones de los cuadros amarillos. Los primeros dos cuadros de guía te permitirán conocer mejor tanto el programa como la estructura. Los siguientes te permitirán representar partes importantes de la molécula. Deberás adjuntar y describir en el formulario de resultados algunas de las imágenes que crees. Si tienes dificultades visualiza las VideoGuias que encontrarás en cada parte.
Si no has abierto aún la estructura hazlo de la siguiente forma:
En el menú principal de VMD selecciona File -->New Molecule.... A través de Browse... navega en tu sistema hasta encontrar el fichero pdb que has descargado. Selecciónalo, y abrelo con OK y después Load.
Para continuar abre la ventana de Representaciones (Graphics -->Representations)...
En la estructura hay cuatro cadenas. De momento vamos a trabajar solo con la cadena A (chain A)...
Recuerda que no todos los átomos son proteicos. Vamos a separar átomos proteicos y no proteicos...
CPK es una representación con bolas y barillas de manera que aparecerán enlaces entre átomos cercanos cuando así corresponda. Ello te permite observar con claridad que los átomos no proteicos son de dos tipos.
Puedes saber fácilmente de qué átomos se trata marcándolos en el Display. Para ello puedes cambiar fácilmente la funcionalidad del ratón (Mouse -->Label -->Atoms). Puedes hacer lo mismo de manera más rápida de la manera siguiente:
Para entender mejor la estructura de la proteína vamos a alargar la cadena poco a poco.
Esconde la representación que tengas visible (click dos veces sobre la linea de la representación) y crea una nueva (Create Rep)
Cuando logres visualizar el primer residuo, puedes ver su nombre y la denominación de sus átomos seleccionándolos con el ratón:
Continúa alargando la cadena...
Quizá veas que la estructura que aparece es bastante regular. Para observar mejor como es, duplica la representación de la forma siguiente:
La estructura secundaria también puede evaluarse a través del diagrama de Ramachandran, que podemos obtener utilizando comandos especiales de análisis que se encuentran en el menú de Extensions.
Ahora representaremos con detalle el grupo hemo y el O2 en una de las cadenas de la hemoglobina. En la representación destacaremos también dos residuos importantes de la proteína: las histidinas histidinas proximal y distal. Para utilizar el grupo hemo y el oxígeno te será de utilidad recordar sus denominaciones como residuo en el fichero pdb (Parte 1.4). Sigue la guía del cuadro:
Recuerda que puedes combinar varias representaciones si necesitas utilizar estilos diferentes.
La histidina proximal es la que se encuentra coordinada al Fe2+ del grupo hemo. La distal es la siguiente más cercana, por la cara opuesta del plano del hemo. Para representarlas "a parte" necesitamos conocer su numeración de residuo:
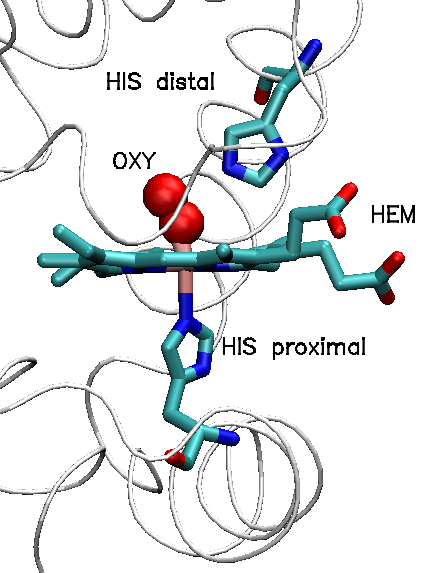
Si quieres completar tu representación con mas residuos cercanos al sitio de unión de oxígeno, puedes tomar el átomo de Fe como referencia y buscar otros átomos cercanos a él:
Para terminar estudiaremos la proteína completa, incluyendo sus cuatro subunidades. Para este tipo de representaciones de estructura cuaternaria conviene utilizar visualizaciones de superficie, que nos dan una idea clara de la forma de la molécula y permiten analizar las interacciones entre subunidades. Dichas superficies pueden colorearse según el tipo de cadena, el tipo de residuo, etc.
Esconde tus representaciones activas (click dos veces sobre la linea de la representación); trabajaremos con otras nuevas.
Ahora cada cadena se muestra con un color diferente. Recuerda que las cadenas son iguales dos a dos. Las cadenas A y C son de tipo α. Las cadenas B y D son de tipo β. Con las cuatro cadenas visibles puedes ver que las uniones entre ellas no son en todos los casos igual de cercanas. Prueba a representarlas en pares (α1 con β1, α1 con β2, α1 con α2, β1 con β2,):
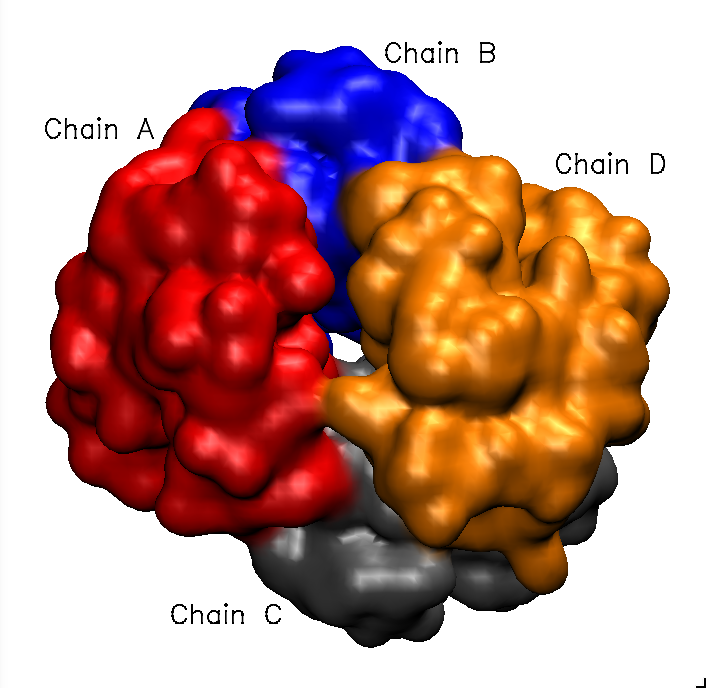
Para ver las características de la superficie de una cadena que interacciona con otra, cambia la representación de una de ellas a un modo esquemático (por ejemplo, Lines) y colorea la superficie de la otra según el tipo de residuo (ResType):
