Importar (y exportar) datos en R
Vicente Coll & Pedro J. Pérez
Junio 2017
1. Intro
Ya sabemos que R es un lenguaje de programación orientado al análisis de datos. Lo primero que tenemos que hacer para empezar un análisis con datos en R es, evidentemente, cargar los datos en R. En realidad en este tutorial aprenderemos a importar y exportar datos en diferentes formatos.
Hay datos de muchos tipos y en muchos formatos: imágenes, texto, … , pero en el curso nos centraremos en conjuntos de datos que pueden almacenarse en hojas de calculo, ya que esta es la forma habitual de trabajar con datos en las ciencias sociales.
Utilizando un diagrama de este fantástico libro, estamos en la casilla de salida de cualquier análisis de datos.
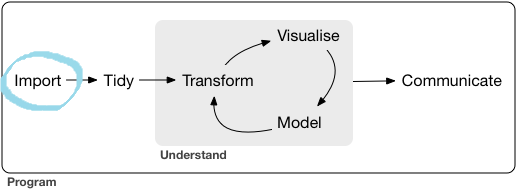
Primera etapa: Importar datos (http://r4ds.had.co.nz/)
Cargar datos es una de las primeras frustraciones de alguien que comienza a aprender R. Generalmente piensan: pero si en Excel/SSPSS sólo tengo que pinchar en el fichero!! Como mucho tengo que usar los menús desplegables!! En R esto también es posible: R tiene 2 formatos de datos propios que se abren simplemente haciendo doble click y la última versión de RStudio también permite cargar datos a través de menús; pero …. no os acordáis de la Investigación Reproducible!!
¿Por qué no usar R-base? [OPCIONAL]
Ya hemos dicho que RStudio carga datos a través de menús, pero no utiliza las funciones de R-base, sino de otros paquetes, concretamente readr y haven
R tiene ya unos 20 años. Las funciones de R-base se construyeron pensando en los estadísticos de hace 20 años (hoy se llamarían analistas de datos). Modificar las funciones de R-base haría que código antiguo dejase de funcionar, así que la mayoría de avances y mejoras se producen en los packages.
Las funciones de readr tratan de ser lo mas parecidas a las funciones equivalentes de R-base pero en cierto sentido mejorándolas y haciéndolas más consistentes; por ejemplo para leer datos CSV la función de R-base es read.csv(); mientas que la función equivalente de “readr” es read_csv(). Las dos hacen lo mismo, leer datos en formato CSV, pero las nuevas funciones tienen algunas ventajas:
Son más rápidas.
Encajan más en el workflow/paradigma de la investigación reproducible. Por ejemplo, algunas de las funciones de R-base heredan algunas opciones del sistema operativo y las variables de entorno, haciendo posible que un script que funciona en un ordenador no funcione en otro. (Esto aún puede pasarnos a nosotros en el curso. Esperemos que no!!).
En lugar de generar data.frames, producen tibbles. Las tibbles son en realidad data.frames pero con algunas particularidades.
Las tibbles o “data frames tuneados” tienen unas ciertas ventajas: no convierten por defecto vectores de texto en factores, no usan row names, ni transforman los column names (estás 3 cosas que sí hacen los data.frame pueden provocar algunas complicaciones, así que mejor tener herramientas que las sorteen).
Obtener información sobre un pkg [BÁSICO-GENÉRICO] (ya se trató en el tutorial nº 2)
Como para importar/exportar datos vamos a usar funciones de varios packages, tenemos que saber como acceder a su documentación.
Generalmente los packages tienen una extensa documentación. Para acceder a la documentación de un pkg podemos hacer lo siguiente:
Ir al repositorio oficial del package (por ejemplo para el pkg “readr” buscando en el navegador “CRAN readr”). Concrétamente, la documentación oficial del package
readrestá en https://cran.r-project.org/web/packages/readr/index.html. Todos los packages en CRAN tienen un Reference Manual: un pdf con documentación extensa de cada una de las funciones del pkg. Habitualmente los packages también tienen unos documentos llamados vignettes que explican de forma más genérica para que sirve y cómo se usa el package.Desde RStudio también podemos acceder a la documentación oficial del pkg
#- abrimos en RStudio la ayuda del pkg readr
help(package = readr)Obtener información de una función [BÁSICO-GENÉRICO]
El paquete readr que vamos a utilizar tiene más de 80 funciones. Muchas, sí, pero solo usaremos algunas y todas tienen una estructura parecida. Por ejemplo usaremos:
read_csv,read_csv2,read_delim,read_table,read_rds….write_csv….
Si queremos ver la documentación de una función concreta; por ejemplo de la función read_csv(), tenemos que utilizar help(). Veámoslo:
#- visualiza la documentación de la función read_csv del package readr.
help(read_csv, package = "readr")
#- así tambien funcionaría. Si hubiese ambigüedad, RStudio nos avisaría
help(read_csv)Un truco muy útil es este atajo de teclado: presionando F1 cuando estás sobre el nombre de una función llama a help() y entonces muestra la documentación de la función. Esta forma de obtener ayuda de una función es mucho más cómoda.
¿Cómo saber a que package pertenece una función?
A veces es conveniente saber que package contiene una función. Podemos hacerlo con:
#- usamos la función"find" para encontrar en que package esta la función "help"
find("help")## [1] "package:utils"Datos precargados en R [OPCIONAL]
R-base viene con muchos datos precargados; concretamente en el pkg de R-base llamado datasets. Además muchos packages contienen también conjuntos de datos. Para ver los datos que tenemos precargados y disponibles en R se usa la función data():
#- se abrirá una ventana con el listado de datos disponibles
data()
#!! guardamos el listado de datos en un data.frame llamado "aa"
aa <- as.data.frame(data()[[3]]) Si queremos ver los datos que hay en un package concreto usaremos data(package = "pkg_name")
#- vemos en una ventana el listado de datos disponibles en el pkg ggplot2
data(package = "ggplot2")
#!! guardamos el listado de datos del pkg ggplot2 en el df "aa"
aa <- as.data.frame(data(package = "ggplot2")[[3]]) %>% select(-2)
#!! guardamos el listado de datos del pkg ggplot2 en una tibble
aa <- as_tibble(data(package = "ggplot2")[[3]]) %>% select(-2) Por ejemplo, el package ggplot2 tiene los siguientes conjuntos de datos:
| Package | Item | Title |
|---|---|---|
| ggplot2 | diamonds | Prices of 50,000 round cut diamonds |
| ggplot2 | economics | US economic time series |
| ggplot2 | economics_long | US economic time series |
| ggplot2 | faithfuld | 2d density estimate of Old Faithful data |
| ggplot2 | luv_colours | ‘colors()’ in Luv space |
| ggplot2 | midwest | Midwest demographics |
| ggplot2 | mpg | Fuel economy data from 1999 and 2008 for 38 popular models of car |
| ggplot2 | msleep | An updated and expanded version of the mammals sleep dataset |
| ggplot2 | presidential | Terms of 11 presidents from Eisenhower to Obama |
| ggplot2 | seals | Vector field of seal movements |
| ggplot2 | txhousing | Housing sales in TX |
Podemos ver todos los datasets que hay en los packages que tenemos en nuestra librería de packages de nuestro ordenador:
# !! abre una ventana donde se ve el listado de todos los datasets que contienen los packages de nuestra librería
data(package = .packages(all.available = TRUE))2. Tipos de datos que veremos
Introduciremos funciones para importar/exportar datos de los siguientes formatos:
Datos en formato texto (o tabulares)
- CSV:
.csv(comma separated values o , en castellano, datos separados por comas) - otros datos en formato texto
- CSV:
Formatos de otros programas (software propietario)
- EXCEL:
.xlsy.xlsx - SPSS:
.savy.por - STATA:
.dta - SAS:
.sas
- EXCEL:
Formatos propios de R
- R objects:
.RDatao.rda - Serialized R objects:
.rds
- R objects:
Otros Formatos
- JSON
- XML
Además aprenderemos como bajar datos a través de APIs:
- Eurostat
- INE
- Banco Mundial
Estrategia que seguiremos para aprender a Importar/Exportar datos
Lo que vamos a hacer en este tutorial para aprender a importar (y exportar) datos en R es elegir un fichero de datos precargado en R y exportarlo a un determinado formato para luego importar el archivo generado o exportado. Repetiremos esto para distintos formatos de datos.
Da igual que archivo de datos usar, pero por fastidiar a Vicente (y porque es un fichero ligero) utilizaremos un conjunto de datos famoso: “el iris dataset” que fue utilizado por Ronald Fisher. Iris contiene 150 observaciones de 5 variables: mediciones de 5 características sobre 150 flores de la especie Iris.
¿Cómo podemos ver que variables (y de que tipo) hay en un df?
Supongamos que ya hemos cargado un conjunto de datos y que está almacenado en un df, ¿cómo podemos ver que variables (y de que tipo) hay en el df?
Vamos a ver los nombres de las variables (columnas) del dataset iris:
# names() muestra los nombres de las variables de un dataframe
names(iris)## [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width"
## [5] "Species"No nos hace falta, pero veamos los primeros valores de iris:
# head() muestra las n (ne este caso 4) primeras filas de un dataframe
head(iris, n = 4)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosaLa función summary(), !que casualidad!, nos hace un resumen (!) del df
# Fijate que la variable "Species" no tiene media, ni minimo, ni max. ... es porque es un factor
summary(iris)## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
## 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
## Median :5.800 Median :3.000 Median :4.350 Median :1.300
## Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
## 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
## Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
## Species
## setosa :50
## versicolor:50
## virginica :50
##
##
## SIEMPRE-SIEMPRE hay que chequear de que clase son las variables que contiene el df.
#- ver la estructura del df. Visualizaremos los nombres y el tipo de las variables
str(iris)## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...También podéis usar la función names_v_df_pjp() del package personal.pjp. El package no está en CRAN, está alojado en Github
#(!!) instalo y cargo el pkg personal.pjp
if (!require(devtools)) {install.packages("devtools")}
if (!require(personal.pjp)) {devtools::install_github("perezp44/personal.pjp")}
library("personal.pjp")
aa <- names_v_df_pjp(iris)
aa## variable q_zeros p_zeros q_na p_na type unique min max mean sd
## 1 Sepal.Length 0 0 0 0 numeric 35 4.3 7.9 5.84 0.83
## 2 Sepal.Width 0 0 0 0 numeric 23 2.0 4.4 3.06 0.44
## 3 Petal.Length 0 0 0 0 numeric 43 1.0 6.9 3.76 1.77
## 4 Petal.Width 0 0 0 0 numeric 22 0.1 2.5 1.20 0.76
## 5 Species 0 0 0 0 factor 3 NA NA NA NAVamos YA a exportar (e importar) “iris” a diferentes formatos. Empezamos!!
3. Datos tabulares (o de texto)
Estamos acostumbrados a visualizar datos en formato tabular; es decir, como una tabla. Generalmente las columnas son variables y las filas son observaciones de esas variables para diferentes unidades de análisis (“individuos”).
Las columnas se separan con un carácter (generalmente la coma) y las filas con un salto de linea.
Podemos pensar que dependiendo de como se separen las observaciones tenemos distintos tipos de datos tabulares, pero en realidad su estructura es similar: variables en columnas y las observaciones de un individuo separadas por una marca o carácter. Este carácter puede ser un espacio, un tabulador, una coma, punto y coma etc… El formato tabular mas extendido es el CSV, donde las observaciones están separadas por comas.
Estos datos se pueden visualizar en los editores de texto y por eso también se llaman datos en formato texto.
Podemos pensar que hay 2 grupos de datos tabulares:
- delimitados por caracteres
- de anchura fija
El package readr lee datos tabulares con las siguientes funciones:
- si los datos están delimitados por caracteres utiliza:
read_delim(),read_csv(),read_tsv()… - si los datos son de anchura fija:
read_fwf()yread_table()
Sólo veremos como importar/exportar datos tabulares del primer tipo; es decir, separados por caracteres. Comenzaremos con el formato CSV que es el más utilizado.
CSV
CSV significa “comma separated data”. En realidad CSV es un caso particular de “tabular o text data”
Recordad que tenemos que exportar el dataframe iris a formato CSV y luego importarlo.
Para exportar iris a un fichero en formato CSV utilizaremos la función write_csv(): solo hay que decirle el objeto que queremos exportar (en este caso un df “iris”) y el nombre (junto con la ruta) del archivo donde queremos guardarlo.
Podemos especificar la ruta completa. Por ejemplo:
#- exporta en formato CSV el df iris al fichero "iris.csv"
#- Cuidado!! es una ruta absoluta. No funcionará en todos los ordenadores
write_csv(iris, file = "C:/Users/perezp/Desktop/iris.csv")En realidad no hace falta especificar la ruta completa. Si solo especificamos el nombre del archivo, R lo guardará en el directorio de trabajo.
#- exporta en formato CSV el df iris al fichero "iris.csv". Como no se especifica la ruta, se grabará en el directorio de trabajo
write_csv(iris, file = "iris.csv")Recuerda que para saber cual es tu directorio de trabajo puedes usar la función getwd() y puedes cambiarlo desde los menús de RStudio o con setwd(). Por ejemplo:
#- almacenamos en el objeto "path_wd" la ruta de mi directorio de trabajo
path_wd <- getwd()
#- Podemos fijar fijamos el directorio de trabajo donde queramos. Por ejemplo (en tu ordenador no funcionará):
setwd("C:/Users/perezp/Desktop/Mis_datos/")Recomendamos trabajar con Rprojects y guardar los ficheros de datos en una carpeta llamada /datos/.
Por lo tanto, para exportar los datos de “iris” en la subcarpeta /datos/ dentro del proyecto, hay que hacer lo siguiente:
#- exporta en formato .csv el df my_iris al fichero "iris.csv". Se guardará en la subcarpeta "datos" del proyecto
write_csv(iris, "./datos/iris.csv")Si queremos, podemos poner explícitamente los argumentos (o parámetros) de la función write_csv():
#- Otra vez exportamos en formato .csv el df iris. Esta vez explicitamos las opciones o parámetros de la función
write_csv(iris, path = "./datos/iris.csv", col_names = TRUE)Bien, ya hemos exportado “iris” a un fichero en formato CSV, ahora vamos a importarlo.
Para importar los datos del fichero “iris.csv” hacemos lo siguiente:
#- importamos los datos del fichero "iris.csv" y los guardamos en un objeto que llamamos "iris_imp_csv". Recuerda que acabamos de exportar "iris" a la carpeta "/datos/" dentro del Rproject
iris_imp_csv <- read_csv("./datos/iris.csv")Así de sencillo!! Además la mayoría de programas permiten leer y exportar datos en CSV; así que si trabajamos con otro software (Excel, SPSS …), siempre podemos pasar nuestros datos a R exportándolos a CSV; y desde R podemos hacer lo mismo.
Algunas opciones de read_csv() que conviene conocer
A veces los datos tienen ciertos problemas que hay que arreglar; por lo que conviene conocer algunas opciones de read_csv():
col_names: read_csv() asume que la primera fila contiene los nombres de las variables. Esto puede cambiarse con
col_names = FALSE. Puedes proveer nombres a las variables (o columnas) concol_names = c("X1", "X2")skip:read_csv() por defecto importa todas las filas del archivo, pero puedes hacer que comience a importar en la fila que quieras con
skip = nna: En algunos ficheros con datos tabulares los NAs se especifican con algún carácter. Esto podemos tratarlo al leer los datos con la el argumento
na = "xxx"
Por ejemplo, el chunk que ves abajo utiliza read_csv() para cargar el fichero “my_fichero.csv”. Comienza a importar datos desde la quinta columna, trata los valores -44 y $ como NAs y provee un vector con los nombres que queremos para las variables (o columnas)
mis_datos <- read_csv("my_fichero.csv", skip = 5, na = c("-44", "$"), col_names = c("X1", "X2", "YY", "X4", "ZZ"))Otros datos tabulares
En realidad, todos los datos tabulares (separados por caracteres) son muy similares. ¡Solo se diferencian en el carácter que hace de separador.
El package “readr” tiene una función especifica para cada tipo de datos tabulares. Por ejemplo, si el separador es un punto y coma, la función para importar estos datos es read_csv2(); si el separador es un tabulador, la función es read_tsv(). Pero también tiene una función genérica que sirve para cualquier tipo de separador: read_delim(). Obviamente usaremos esta función.
Por ejemplo, podemos cargar el fichero “my_iris_exportado.csv” que hemos exportado anteriormente utilizando la función genérica read_delim(), solo hay que decirle que el separador es una coma. Se lo decimos con la opción delim = ",". Veámoslo:
#- importamos los datos del fichero "iris.csv" y los guardamos en un objeto que llamamos iris_imp_csv_2. Fíjate en el argumento 'delim'
iris_imp_csv_2 <- read_delim("./datos/iris.csv", delim = ",")Como el formato tabular mas extendido es el CSV; en general, no tendremos necesidad de exportar datos tabulares separados por caracteres distintos a la coma, pero si quisiéramos hacerlo, podríamos hacerlo con write_tsv() o con write_delim():
#- exportamos iris en formato tabular separado por punto y coma.
write_delim(iris, "./datos/iris_2.txt", delim = ";")
#- exportamos iris en formato tabular separado por tabuladores
write_delim(iris, "./datos/iris_3.txt", delim = "\t")
#- exportamos iris en formato tabular separado por un espacio en blanco
write_delim(iris, "./datos/iris_4.txt", delim = " ")Si quisiéramos importarlos, tendríamos que hacer:
#- exportamos iris en formato tabular separado por punto y coma.
read_delim("./datos/iris_2.txt", delim = ";")
#- exportamos iris en formato tabular separado por tabuladores
read_delim("./datos/iris_3.txt", delim = "\t")
#- exportamos iris en formato tabular separado por un espacio en blanco
read_delim("./datos/iris_4.txt", delim = " ")
4. Formatos propietarios
Hasta que R haga desparecer a SPSS ….. ;) aún será necesario importar algún fichero en formatos de software propietario como los de Excel, SAS, Stata , SPSS y Eviews
Normalmente necesitaremos importar datos en esos formatos, solo alguna vez tendremos que exportarlos, ya que todos estos programas pueden leer datos CSV.
Lo que sí puede resultarnos útil es exportar datos en formato .xls, ya que Excel es muy útil para visualizar datos en formato tabular y también porque mucha gente prefiere (o solo sabe) leer/analizar datos en Excel.
Excel
Exportar a excel
Hay varios packages que graban datos en formato .xls. Pero el más sencillo es el package xlsx. Veámoslo:
# library(xlsx)
write.xlsx(iris, "./datos/iris.xlsx")La función write.xlsx() permite especificar el nombre del libro y alguna cosa más; por ejemplo:
# library(xlsx)
write.xlsx(iris, "./datos/iris.xlsx", sheetName = "IRIS", row.names = FALSE)La función write.xlsx() permite añadir datos a un archivo .xlsx preexistente; para ello tenemos que usar la opción append = TRUE:
# library(xlsx)
write.xlsx(iris, "./datos/iris.xlsx", sheetName = "IRIS_2", append = TRUE)Importar archivos en formato excel
También hay varios paquetes, pero usaremos el mismo que usa RStudio en sus menús: readxl
readxl permite leer ficheros .xls y .xlsx. Por ejemplo:
# library(readxl)
iris_imp_xls <- read_excel("./datos/iris.xlsx")Podemos especificar el libro que queremos abrir, ya sea especificando su nombre o su posición en el fichero
# library(readxl)
iris_imp_xls <- read_excel("./datos/iris.xlsx", sheet = 2)
iris_imp_xls <- read_excel("./datos/iris.xlsx", sheet = "IRIS_2")La función read_excel() tiene más posibilidades; como ejemplo, la opción skip = 4 permite empezar a importar a partir de la cuarta fila.
Si queremos importar todos los libros (o sheets) de un archivo Excel, podemos hacerlo así:
# library(readxl)
# (!!!)
IRIS_list <- lapply(excel_sheets("./datos/iris.xlsx"), read_excel, path = "./datos/iris.xlsx")Hemos guardado los 2 sheets del archivo “./datos/iris.xlsx” en un objeto R llamado IRIS_list. Este objeto es una lista con 2 elementos. cada elemento contiene los datos de cada uno de los 2 sheets. Podemos verlo con str():
# (!!!)
str(IRIS_list)## List of 2
## $ :Classes 'tbl_df', 'tbl' and 'data.frame': 150 obs. of 5 variables:
## ..$ Sepal.Length: num [1:150] 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## ..$ Sepal.Width : num [1:150] 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## ..$ Petal.Length: num [1:150] 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## ..$ Petal.Width : num [1:150] 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## ..$ Species : chr [1:150] "setosa" "setosa" "setosa" "setosa" ...
## $ :Classes 'tbl_df', 'tbl' and 'data.frame': 150 obs. of 6 variables:
## ..$ NA : chr [1:150] "1" "2" "3" "4" ...
## ..$ Sepal.Length: num [1:150] 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## ..$ Sepal.Width : num [1:150] 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## ..$ Petal.Length: num [1:150] 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## ..$ Petal.Width : num [1:150] 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## ..$ Species : chr [1:150] "setosa" "setosa" "setosa" "setosa" ...Si quisiéramos recuperar los datos en el formato en el que estamos habituados (dataframes) lo haríamos así:
# (!!!)
primer_iris <- IRIS_list[[1]]
segundo_iris <- IRIS_list[[2]]Otros formatos propietarios
Veremos SPSS, Stata y SAS.
Utilizaremos el mismo package que usa RStudio: haven. Si necesitásemos importar otro tipo formato es muy posible que se pueda hacer con los los packages foreign y rio
SPSS
Exportación a SPSS (formato .sav)
# library(haven)
write_sav(iris, "./datos/iris.sav")Importacion de ficheros .sav (tb .por)
# library(haven)
iris_imp_spss <- read_spss("./datos/iris.sav")Stata
Exportación a STATA (formato .dta)
Se puede exportar con haven (pero no permite labelled data!!), así que mejor hacerlo esta vez con el package foreign
# library(foreign)
write.dta(iris, "./datos/iris.dta")Importacion de ficheros STATA (.dta)
# library(haven)
iris_imp_stata <- read_stata("./datos/iris.dta")SAS
Exportación a SAS
haven exporta bien a SAS; pero … los nombres de las variables no pueden contener puntos, así que usamos otro fichero de datos mtcars. Podríamos haber cambiado el nombre de las columnas de iris (quitando los puntos), pero lo dejamos para el próximo tutorial.
# mtcars es un dataset del pkg ggplot2, asi que ggplot2 debe estar cargado
# library(ggplot2)
# library(haven)
write_sas(mtcars, "./datos/mtcars.sas") Importacion de ficheros SAS
# library(haven)
mtcars_imp_sas <- read_sas("./datos/mtcars.sas")5. Formato(s) propios de R
Guardar datos en formatos como txt, csv o Excel es lo más habitual si quieres abrir estos datos en otros programas; pero al grabar en estos formatos guardas los datos, PERO no guardas la estructura de los datos; es decir, si por ejemplo una columna la has definido como un factor o como integer, esta información se perderá. En estos casos, una solución es usar el formato propio de R.
Hay dos posibilidades:
- si quieres grabar un solo objeto, es preferible hacerlo como
Rds - si quieres grabar varios objetos tienes que hacerlo como
RDatao abreviado comoRda
RData
RData (o Rda) es un formato especifico de R, pero tiene dos ventajas:
- es bastante eficiente
- permite guardar varios objetos en un único archivo
Para exportar my_iris a un fichero en formato .RData utilizaremos la función save(): solo hay que decirle el objeto que queremos exportar (en este caso un df/tibble llamado “my_iris”) y el nombre (junto con la ruta) del archivo donde queremos guardarlo.
El formato .RData tienen la ventaja de que puedes guardar varios objetos a la vez.
save(mtcars, iris, file = "./datos/mtcars_and_iris.RData")Incluso puede guardar todos los objetos de la sesión. Mejor no hacerlo
save(list = ls(all = TRUE), file= "./datos/all_objects.RData")O todo el espacio de trabajo:
save.image(file = "./datos/my_work_space.RData")para luego cargarlos con:
load("./datos/all_objects.RData")
load("./datos/my_work_space.RData")Para cargar o importar datos en formato .RData basta con arrastrarlos dentro de RStudio, o seguir la ruta de menús File > Open File pero para hacerlo en un script se hace así:
Un posible pega es que al cargar datos con load() se cargan todos los datos que guardaste y además se cargan en el espacio de trabajo con el mismo nombre con que los guardaste. Esto puede ser una pega pues es posible que haya un objeto que se llame igual que un objeto con el que estás trabajando en RStudio.
RDS (Serialized R objects)
Una “desventaja” del formato RData es que al importar un fichero .RData, los objetos que contiene se cargan siempre con el nombre con el que fueron grabados. ¿Qué más da? Bueno, puede parecer que no es muy importante, pero a veces esto puede limitar el workflow, así que muchas veces es preferible grabar como .RDS con la función write_rds(). La pega es que está función solo permite guardar un objeto.
Para exportar iris a formato RDS hacemos:
write_rds(iris, "./datos/iris.rds")Para leer o importar datos RDS (hay que asignarle un nombre, que puede ser o no el nombre original) hacemos:
iris_imp_rds <- readRDS("./datos/iris.rds")6. Otros formatos
Los formatos más frecuentes (al menos en nuestra área) continúan siendo los .csv, .xls, … PERO existen muchos otros formatos.
Hay 2 formatos que es bueno, al menos, saber que existen, porque cada vez son más frecuentes:
- JSON (JavaScript Object Notation)
- XML (Extensible Markup Language).
Para JSON, se recomienda usar el paquete jsonlite y para XML, xml2.
7. Descargar datos de internet
Hay muchísimos datos en internet para descargar; siempre podemos descargarlos usando el navegador, PERO la filosofía del curso es (si podemos) hacerlo todo desde R/RStudio
Desde RStudio, podemos descargar datos con las mismas funciones que usábamos para cargar en el entorno de trabajo los datos que teníamos en nuestro PC. La única diferencia consiste en que, en lugar de proporcionar la ruta al fichero, tendremos que proporcionar la ruta de internet. Por ejemplo:
# cargamos los datos del fichero "bio260-heights.csv"
url <- "https://raw.githubusercontent.com/datasciencelabs/data/master/bio260-heights.csv"
datos <- read_csv(url)A veces podemos necesitar hacer una copia de los datos a nuestro ordenador. En este caso, lo que yo haría es cargar los datos y luego exportarlos a .rds; pero también podemos hacerlo directamente con la función download.file():
# desargamos y almacenamos en nuestro PC los datos del fichero "bio260-heights.csv"
url <- "https://raw.githubusercontent.com/datasciencelabs/data/master/bio260-heights.csv"
destino <- "./datos/bio260-heights.csv"
download.file(url, destino)
dat <- read.csv(destino)A veces, la función de R-base download.file puede tener problemas si el protocolo es https. En estos casos, la función dowload() del pkg dowloader puede solucionarlo:
#install.packages("downloader")
library(downloader)
url <- "https://raw.githubusercontent.com/datasciencelabs/data/master/bio260-heights.csv"
filename <- basename(url)
destino <- paste0("./datos/",filename)
download(url,destino)
dat <- read.csv(destino)8. Web Scrapping
El proceso y acciones para recopilar información de la Web se conoce como web scrapping. Este proceso se puede hacer manualmente, pero lo habitual es automatizarlo utilizando software. Se puede acceder a los datos directamente pero actualmente es muy común hacerlo a través de APIs, ya que la mayoría de organismos/empresas tienen una o varias APIs.
API significa “Aplication Programming Interface” y se puede entender como un mecanismo que nos permite interactuar (por ejemplo para hacer una petición de datos) con un servidor de internet. Por ejemplo, muchos bancos tienen APIs a las que se les pueden hacer peticiones, esto hace posible que se desarrollen apps para hacer ciertas operaciones bancarias; es decir, una API es un mecanismo que nos permite acceder y/o interactuar con determinadas funciones de un servicio web.
Las APIs facilitan mucho la recopilación de datos al poderse acceder a ellas de forma programática ya que proveen de un proceso de acceso a ellos estandarizado: se envía una “http request” a la API y se reciben los datos en un determinado formato, generalmente JSON o XML.
En el entorno R se pueden desarrollar paquetes para acceder a APIs; por ejemplo, vamos a utilizar el paquete de R eurostat para acceder a la API de Eurostat y descargar datos directamente en R. Veámoslo:
Eurostat
Eursotat tiene una API que permite hacer peticiones de datos. Obviamente, para poder hacer peticiones de datos a través de su API has de conocer su sintaxis; si estás interesado puedes empezar aquí. Nosotros accederemos a Eurostat a través del package eurostat. Si estas interesado en bajar datos de Eurostat es conveniente que uses esta vignette y la cheat sheet. Veamos un ejemplo:
# install.packages("eurostat")
library("eurostat")
#------------------ buscamos "un tema" con search_eurostat
aa1 <- search_eurostat("employment", type = "all")
#------------------ elegimos una tabla de Eurostat
my_table <- "cult_emp_sex" #- elegimos una tabla; por ejemplo "cult_emp_sex": empleo cultural por genero"
label_eurostat_tables(my_table) #- da informacion sobre la Base de datos q estas buscando
#------------------ descargamos los datos con get_eurostat()
df <- get_eurostat(my_table, time_format = 'raw', keepFlags = T ) #- bajamos los datos de una tabla
df_l <- label_eurostat(df) #- pone labels: Spain en lugar de su código (mas legible,menos facil de programar)
#------------------ los arreglamos un poco
library("personal.pjp")
aa <- val_unicos_df_pjp(df_l) #- ver los valores unicos de cada columna
df_code <- label_eurostat(df, code = c("geo", "unit"))INE
¿El INE tiene API? Pues sí, aquí puedes “verla”, pero …
Hace poco tuvimos que utilizar datos del INE y, en lugar de usar la API, nos bajamos los datos así:
library("pxR") #- para trabajar con datos PC-Axis
library("tidyverse")
library("personal.pjp")
file_name <- "http://www.ine.es/jaxiT3/files/t/es/px/4189.px?nocab=1"
df <- read.px(file_name) %>% as.data.frame %>% as.tbl
aa <- val_unicos_df_pjp(df) #- ver los valores unicos de cada columnaBanco Mundial
Para acceder a la API del Banco Mundial hay, actualmente, 2 paquetes de R: WDI y wbstats.
Podemos bajar datos del Banco Mundial con el paquete WDI así:
install.packages("WDI")
library("WDI")
#---- buscamos datos relacionados con GDP
aa <- WDIsearch('gdp')
aa <- WDIsearch('gdp.*capita.*constant')
#---- descargamos "NY.GDP.PCAP.KD": GDP per capita (constant 2010 US$)
df <- WDI(indicator = "NY.GDP.PCAP.KD")
#---- podemos filtrar la querry
df <- WDI(indicator = "NY.GDP.PCAP.KD", country = c('MX','CA','US'), start = 1960, end = 2017)Podemos bajar datos del Banco Mundial con el paquete wbstats así:
install.packages("wbstats")
library("wbstats")
#------- lista de indicadores disponibles
aa <- wb_cachelist
#---- buscamos datos relacionados con GDP
aa <- wbsearch(pattern = "gdp")
aa <- wbsearch('gdp.*capita.*constant')
#---- descargamos "NY.GDP.PCAP.KD": GDP per capita (constant 2010 US$)
df <- wb(indicator = "NY.GDP.PCAP.KD")
#---- podemos filtrar la querry
df <- wb(indicator = "NY.GDP.PCAP.KD", country = c('MX','CA','US'), startdate = 2000, enddate = 2017)Aquí tenemos un post en el que se usa el pkg wbstats para luego graficarlos con otro paquete que permite accader al API de Google Visualisations:
suppressPackageStartupMessages(library(googleVis))
library(wbstats)
library(data.table)
library(googleVis)
# Download World Bank data and turn into data.table
myDT <- data.table( wb(indicator = c("SP.POP.TOTL", "SP.DYN.LE00.IN", "SP.DYN.TFRT.IN"), mrv = 60))
# Download country mappings
countries <- data.table(wbcountries())
# Set keys to join the data sets
setkey(myDT, iso2c)
setkey(countries, iso2c)
# Add regions to the data set, but remove aggregates
myDT <- countries[myDT][ ! region %in% "Aggregates"]
# Reshape data into a wide format
wDT <- reshape(
myDT[, list(country, region, date, value, indicator)], v.names = "value", idvar=c("date", "country", "region"),
timevar="indicator", direction = "wide")
# Turn date, here year, from character into integer
awDT <- wDT[, date := as.integer(date)]
setnames(awDT, names(awDT), c("Country", "Region","Year", "Population", "Fertility", "LifeExpectancy"))
#- GoogleVis graphic
M <- gvisMotionChart(wDT, idvar = "Country", timevar = "Year", xvar = "LifeExpectancy",
yvar = "Fertility", sizevar = "Population", colorvar = "Region")
# Ensure Flash player is available an enabled
print(M,'chart')CrossRef
El paquete rcrossref permite acceder a varias de las APIs de CrossRef. ¿Que qué es CrossRef? Pues es un servicio que permite, entre otras cosas, facilitar el proceso de referenciar artículos en tus papers. Aquí lo explican. Hay otro package para acceder a CrossRef: crminer este pkg permite bajarse el texto del documento, pero claro, el texto ha de estar disponible!!
install.packages("rcrossref")
library("rcrossref")
#----- con cr_cn() podemos ver como se cita un determinado articulo en un determinado formato, por ejemplo "apa"
my_doi <- "10.1111/j.1467-6486.2012.01072.x"
cr_cn(dois = my_doi, format = "text", style = "apa")
cr_cn(dois, format = "bibtex", style = "apa", locale = "en-US",
raw = FALSE, .progress = "none", ...)
#------ con cr_citation_count() ùedes ver el numero de citas de un artículo/DOI
aa <- cr_abstract(doi = my_doi)
#------ con cr_abstract()
aa <- cr_abstract(doi = "10.1109/TASC.2010.2088091")
#------ con cr_journals() vemos journals
aa <- cr_journals(query = "economics", limit = 100) %>% .$data %>% as.tibble()
#------ mucha informacion del articulo
aa <- cr_works(dois = my_doi) %>% .$data %>% as.tibble()Otros pkg for APIs
Hay muchos otros paquetes de R hechos para acceder a APIs (twitter, ECB, spotify, pdfetch, naturalearth, ….). Puedes ver algunos aquí y aquí
Una de las ultimas que he visto ha sido el pkg spooc. En su vignette nos dicen que se pueden acceder a un conjunto de paquetes que contienen: “some form of biodiversity or taxonomic data. Since several of these datasets have been georeferenced, it provides numerous opportunities for visualizing species distributions”
Scrapping tables
Además de utilizar paquetes para acceder a servicios web a través de sus APIs, podemos usar otros paquetes (principalmente rvest) para hacer web scrapping. Puedes ver ejemplos aquí, aquí o aquí
Aquí tenéis un ejemplo sencillo para bajar datos de jurgol:
library(XML)
url <- "http://www.comuniazo.com/comunio/jugadores"
jugadores <- readHTMLTable(url, stringsAsFactors = T, colnames = c("Posicion","Equipo","Jugador","Puntos","Media","Puntos_Casa","Media_Casa","Puntos_Fuera","Media_fuera", "Valor"), colClasses = c("character","character","character","FormattedNumber","FormattedNumber","FormattedNumber","FormattedNumber","FormattedNumber","FormattedNumber"))
aa <- jugadores[[1]] %>% as.tibble()Bibliografía
This R Data Import Tutorial Is Everything You Need. Así es como se llama este tutorial de Datacamp. Es sencillo y no muy largo. Tiene muy buena pinta. En general utiliza funciones de base-R. Lo han actualizado hace 4-5 días. No he tenido tiempo de ver que ha cambiado, pero seguro que a mejor.
La cheat Sheet de data import de RStudio. Muy buen resumen/chuleta
La sección de importar datos de Quick-R. Conciso pero muy claro. Quick-R es muy buen recurso para aprender R.
Un post sobre APIs y R. Supongo que complicado.
Un post que hace un poquito de web scrapping. También puede que complicado, pero es un ejemplo de análisis de datos (completo) con R.