Desenvolupen una IA per a llegir el ‘codi’ dels virus bacterians i dissenyar tractaments amb fags personalitzats
Un equip d’investigació de l’Institut de Biologia Integrativa de Sistemes (I²SysBio), centre mixt de la Universitat de València i del Consell Superior d’Investigacions Científiques (CSIC), ha desenvolupat un innovador sistema d’intel·ligència artificial per a predir quins bacteris poden ser atacats per virus bacterians (fags) en funció de la seqüència d’un enzim clau: la depolimerasa. L’estudi ha sigut publicat en la revista Nature Communications.
La resistència als antibiòtics dificulta cada vegada més el tractament d’infeccions bacterianes. Els fags, que ataquen bacteris, es presenten com a alternativa al tractament antibiòtic normal. No obstant això, identificar quin fag és eficaç per a cada bacteri és complex. Aquest estudi, liderat per Robby Concha-Eloko, Beatriz Beamud, i Pilar Domingo-Calap i Rafael Sanjuán (professora i professor de Genètica de la Universitat de València), proposa l’ús d’intel·ligència artificial per a facilitar aquest procés de predicció.
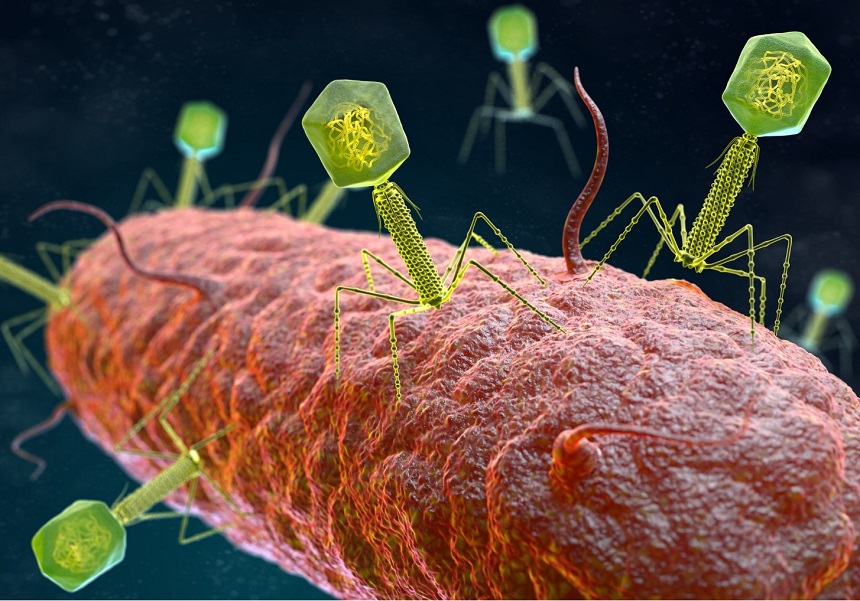
Per a l’elaboració del model han utilitzat el bacteri Klebsiella, inclòs en la llista de patògens bacterians prioritaris de l’OMS, responsable d’infeccions hospitalàries greus i amb una gran resistència als antibiòtics. Els bacteris Klebsiella estan protegits per càpsules de polisacàrids que impedeixen l’activitat dels antibiòtics, així com l’entrada dels fags. Per a superar aquesta barrera, molts fags produeixen depolimerases, enzims que degraden aquestes càpsules i permeten l’entrada del bacteriòfag perquè infecte el bacteri i contribuir al seu tractament.
No obstant això, l’enorme diversitat genètica d’aquestes càpsules –s’han registrat més de 100 serotips d’aquestes estructures en Klebsiella– ha dificultat la predicció de quin fag pot ser l’indicat per a aconseguir travessar la càpsula i infectar el bacteri. A més, aquesta gran varietat de serotips de càpsules converteix Klebsiella en un model ideal per a estudiar la interacció entre fags i càpsules.
Amb aquest objectiu, l’equip d’investigació ha desenvolupat una eina pionera que aprofita la informació genètica de milers de bacteris Klebsiella i els seus virus “adormits” (pròfags) integrats al seu genoma. Analitzant més de 74.000 pròfags i quasi 20.000 seqüències de depolimerases, els investigadors han creat una base de dades que associa cada enzim amb la mena de càpsula bacteriana que pot degradar.
Mitjançant tècniques avançades d’aprenentatge automàtic i models d’intel·ligència artificial inspirats en el processament del llenguatge natural (similars als que usen els traductors automàtics), han aconseguit predir amb gran precisió el tropisme o l'especificitat de cada depolimerasa, és a dir, quins tipus de càpsula bacteriana pot reconèixer i destruir.
Una solució contra els biofilms
Aquest estudi aporta un avanç clau per a la biotecnologia basada en fags o els seus components, ja que permet predir-ne l'especificitat. Aquest fet és fonamental per a dissenyar futures aplicacions, com per exemple solucions contra el biofilm, l’estructura protectora que formen alguns bacteris per a adherir-se a superfícies i resistir tractaments.
Els biofilms es reconeixen cada vegada més com un obstacle important en el tractament de les infeccions. De fet, s’ha demostrat que estan implicats en la cronicitat de malalties com la fibrosi quística, les ferides cròniques, les infeccions relacionades amb pròtesis o les infeccions del tracte urinari.
“L’ús de depolimerasa, ja siga en combinació amb els tractaments actuals (antibiòtics o pèptids antimicrobians) o potencialment com a potenciador del sistema immunitari, pot abordar els problemes relacionats amb la producció de biofilms, la qual cosa condueix a una disminució del risc de fracàs del tractament”, explica Robby-Concha.
“Davant del mètode tradicional, que es basa en un tediós procés de cerca i assaig de fags per a trobar una depolimerasa eficaç, els models d’intel·ligència artificial ens permeten predir-ne l'especificitat in silico”, afirma l’investigador. En aquest sentit, el mètode demostrat en l’estudi permet la generació de biblioteques de depolimerasa que es poden utilitzar per a extraure l’enzim més eficaç, de manera que s'optimitza la degradació de la càpsula i, posteriorment, del biofilm.
Segons afirma Robby-Concha, un dels desenvolupadors d'aquesta eina pionera, malgrat que s’ha utilitzat Klebsiella com a model, aquesta metodologia es pot utilitzar contra qualsevol altre bacteri productor de càpsules. Això inclou la majoria dels patògens prioritaris recollits per l’OMS.
En síntesi, aquest estudi proposa la resolució de les prediccions d’interacció entre fags i hostes de dues maneres. En primer lloc, aprofitant les dades contingudes en genomes bacterians (profags), la qual cosa permet obtenir importants dades d’entrenament, i, en segon lloc, proposant una arquitectura que permeta entrenar el model amb totes les espècies bacterianes alhora (de manera integradora).
Referència: Concha-Eloko, R., Beamud, B., Domingo-Calap, P. et al. "Unlocking data in Klebsiella lysogens to predict capsular type-specificity of phage depolymerases". Nat Commun 16, 8798 (2025). https://doi.org/10.1038/s41467-025-63861-w
Categories: Investigació a la UV , Internacionalització recerca , Institut de Biologia Integrativa de Sistemes (I2SYSBIO) , Difusió i comunicació científica , Cultura Científica , Recerca, innovació i transferència