29 de abril de 2020
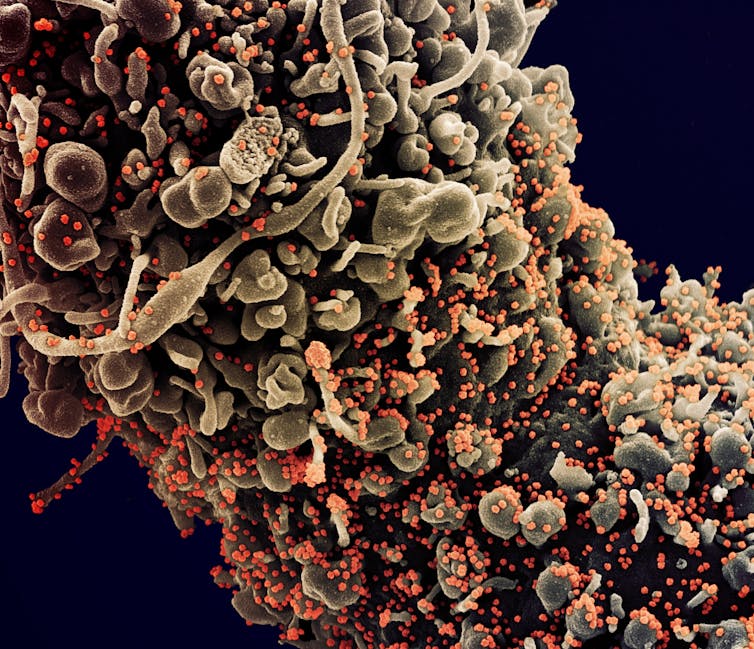
Microfotografía electrónica de barrido coloreada de una célula que muestra signos morfológicos de apoptosis, infectada con partículas del virus del SARS-COV-2 (naranja), aislada de una muestra de un paciente. NIAID, CC BY-SA
Albert Bosch, Universitat de Barcelona; Amelia Nieto, Centro Nacional de Biotecnología (CNB - CSIC); Ana María Doménech Gómez, Universidad Complutense de Madrid; Carlos Briones, Centro de Astrobiología (INTA-CSIC); Covadonga Alonso Martí, Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA); Inmaculada Casas Flecha, Instituto de Salud Carlos III; Javier Buesa Gómez, Universitat de València; Jesús Navas Castillo, Instituto de Hortofruticultura Subtropical y Mediterránea "La Mayora" ( IHSM - CSIC) (EELM-CSIC) ; José Antonio López Guerrero, Universidad Autónoma de Madrid; Josep Quer Sivila, Hospital Universitario Vall d'Hebrón, Generalitat de Catalunya; Juan E. Echevarría Mayo, Instituto de Salud Carlos III y Vicente Pallas Benet, Instituto de Biología Molecular y Celular de Plantas (IBMCP - CSIC)
Para Peter Medawar, premio Nobel de Medicina en 1960, los virus eran un conjunto de “malas noticias envueltas en proteína”. Aunque no responde a ningún criterio científico, esta definición refleja perfectamente la percepción que tenemos de la pandemia del coronavirus SARS-CoV-2 en estos días de confinamiento.
Las “malas noticias” en un virus pueden venir escritas en dos “alfabetos” ligeramente distintos, según su genoma sea de ADN o ARN. Los coronavirus (familia Coronaviridae) constituyen uno de los grupos de virus con el genoma de ARN más largo que se conoce. La información que contiene puede servir para sintetizar al menos 16 proteínas.
Las más esenciales son las que le permiten hacer copias de su propio genoma, las que protegen su ARN y las que le permiten entrar en la célula que va a infectar. Esta última es una proteína que contiene azúcares en su esqueleto y que se proyecta a modo de espículas (proteína S, de spike en inglés) desde la envuelta hacia el exterior. Al microscopio electrónico crean una imagen que recuerda a una corona, de ahí el nombre del virus.
La fidelidad de copia de las moléculas de ARN viral es siempre mucho menor que las de ADN, por lo que los virus de ARN tienden a acumular más mutaciones y adaptarse a nuevos huéspedes con más facilidad que los que poseen un genoma de ADN. Dentro de los virus con genoma de ARN, los coronavirus son una excepción: poseen un sistema de corrección de copia que hace que muestren una menor variabilidad.
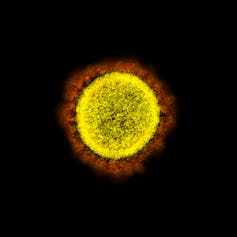
Microfotografía de transmisión de partículas del virus del SARS-CoV-2, aislada de un paciente. NIAID, CC BY-SA
La secuenciación de más 11 000 genomas del SARS-CoV-2 ha puesto de manifiesto que tiene un ritmo de mutación 1 000 veces más lento que el de la gripe o el VIH. Por otro lado, los virus con genoma de ADN son generalmente más difíciles de eliminar porque algunos de ellos pueden producir infecciones latentes o incluso integrarse en el genoma del huésped (esto último también ocurre con los virus de ARN de la familia de los retrovirus).
Los coronavirus humanos se conocen desde hace muchos años y la mayoría de ellos están relacionados con resfriados o afecciones respiratorias leves. En el año 2002 apareció en China el primero letal para los humanos, el SARS-CoV (nombre que proviene de “síndrome respiratorio agudo severo”), que infectó a alrededor de 8 000 personas causando la muerte de 800, lo que supone una tasa de mortalidad del 10 %.
Desapareció en la primavera de 2004 y desde entonces no ha causado más problemas.
El segundo, el MERS-CoV (nombrado por el “síndrome respiratorio de Oriente Medio”), apareció en Arabia Saudí en 2012 y causó aproximadamente el mismo número de muertes, aunque sólo infectó a 2 400 personas. Su tasa de mortalidad fue del 30 %, muy alta, pero lejos de la observada para el virus del Ébola (50 %) y el virus de la rabia (95 %).
Entre los miles de virus que se conocen, solo una pequeña proporción son patógenos en humanos. Las células no tienen una puerta disponible para la entrada de la mayoría de estos microorganismos.
Pero estos diminutos agentes infecciosos han evolucionado mediante altas tasas de mutación y recombinación para utilizar alguna proteína del hospedador como vía de entrada a la célula, donde podrán multiplicarse y perpetuarse: eso es a lo que toda entidad replicativa aspira, y los virus no pueden realizarlo por sí mismos fuera de las células a las que infectan.
Los coronavirus utilizan las proteínas S que forman esas espículas en su exterior para unirse a una proteína enzimática celular llamada ACE2. Este enzima está situado en la superficie de las células de las mucosas, pulmones, arterias, corazón, riñón e intestinos, y tiene la función de regular la presión sanguínea, pero el virus lo emplea como puerta de entrada al contexto celular.
Una vez dentro de su hospedador, el virus campa a sus anchas porque lleva consigo su propia replicasa que le permitirá hacer múltiples copias de su genoma (hasta 100 000 copias en cada célula) y porque el alfabeto de la información que lleva en su ARN es el mismo que utilizamos los humanos.
Los ribosomas celulares no son capaces de identificar ese ARN como extraño. Actúa como un caballo de Troya utilizando la maquinaria celular y traduce dicha información en proteínas del virus. Esto provoca una caída de las funciones esenciales de la célula.
Un origen animal desconocido
El SARS-CoV-2 es de origen animal, aunque en ninguna especie se ha encontrado un coronavirus idéntico o muy similar. Algunas especies de murciélagos y el pangolín malayo, usado como alimento y en medicina tradicional china, son reservorios de virus bastante parecidos, pero los murciélagos estaban hibernando en China en diciembre de 2019, cuando se detectaron los primeros casos en humanos.
Es necesario seguir investigando para identificar las especies animales involucradas en el origen de la pandemia de COVID-19. Los mercados de animales vivos, salvajes y domésticos, en China son muy frecuentes y forman parte de la tradicional cadena alimentaria del país.
Tras esta pandemia, el gobierno chino debería regularizarlos o prohibirlos. De hecho, la siguiente pandemia de virus quizá ya se esté comenzando a fraguar en esos mismos entornos.
Cómo detectar la COVID-19
El diagnóstico de la enfermedad COVID-19, causada por este coronavirus, se lleva a cabo detectando el ARN o los antígenos (proteínas) del virus SARS-CoV-2, o bien los anticuerpos desarrollados por el paciente tras ser infectado.

En el primer caso, las muestras que se utilizan son secreciones respiratorias que pueden contener el virus (exudado nasofaríngeo, exudado nasal, aspirado traqueo-bronquial, esputo), y en ellas se identifica la presencia del ARN viral (mediante su extracción y amplificación por PCR cuantitativa) o de determinados antígenos (empleando sistemas biosensores de distinto tipo).
Por su parte, los anticuerpos del paciente infectado se analizan en sangre, plasma o suero. La detección de anticuerpos frente a SARS-CoV-2 permite conocer qué pacientes están infectados (tras un “período de ventana” de entre 5 y 7 días desde la exposición al virus, que es lo empleado por el organismo en generar la respuesta inmune) y también quiénes han superado la infección incluso sin padecer síntomas.
El SARS-CoV-2 es particularmente contagioso.
Una tasa de letalidad sobreestimada
Los coronavirus se transmiten por aerosoles, donde pueden permanecer viables hasta tres horas, y son estables en diversas superficies: resisten cuatro horas sobre el cobre (monedas), un día entero en el cartón y de dos a tres días en el plástico o el acero inoxidable. El coeficiente de infección R₀ (número promedio de personas a las que cada infectado contagia) es de alrededor de 2,5 (similar a la gripe, pero mucho menos que la viruela o el sarampión, que presentan R₀ de 6 y 15 en promedio) según se ha publicado recientemente en la revista The Lancet, y por tanto su progresión es necesariamente exponencial.
El otro aspecto numérico importante para la progresión de la pandemia es su tasa de letalidad. Para determinarlo de forma fidedigna se requiere realizar un muestreo amplísimo. Los cálculos actuales en base a los datos disponibles dan una tasa de letalidad de alrededor del 7 % de promedio (aunque varían mucho en las distintas franjas de edad: desde un 0,7 % para los menores de 30 años hasta un 20 % para los mayores de 80) en Italia y España.
Esta tasa promedio está sin duda sobrestimada. Tal y como dijo el matemático Andrejs Dunkels, “es fácil mentir con estadísticas” aunque en seguida añadió que “es difícil decir la verdad sin ellas”. Para un cálculo certero habría que conocer el número real de infectados, es decir, hacer los análisis a toda la cohorte (grupo que forma parte de un ensayo clínico, en este caso la totalidad de la población), lo cual es prácticamente irrealizable.
Pero si esa cohorte la tenemos confinada en un crucero, por ejemplo, estamos ante el experimento perfecto. Esto es lo que ocurrió en el Diamond Princess, el que tras un primer positivo se hicieron análisis a los 3 711 pasajeros, incluida la tripulación, y en algunos casos dos veces. Durante las siguientes 4 semanas se detectaron 711 positivos, un 18 % de los cuales fueron asintomáticos.
¿Un millón de infectados?
En un artículo publicado por investigadores del Centro de Modelado Matemático para Enfermedades Infecciosas de Londres, publicado en la revista Eurosurveillance se estimó una tasa de letalidad del 1,1 % en dicho crucero, mucho más baja que el 3,8 % estimado por la Organización Mundial de la Salud (OMS) a nivel global. Si los datos de dicho estudio son correctos y extrapolables, se podría estimar que en España el número de infectados sobrepasaría el millón de personas a día de hoy y esto aumentaría nuestras posibilidades de estar adquiriendo una inmunidad de rebaño que nos proteja para el futuro.
Los coronavirus tienen una membrana lipídica que confiere a la partícula una estabilidad relativamente baja, sobre todo si se la compara con la partícula desnuda del virus del resfriado común. Eso hace que sea relativamente fácil eliminarlos de las manos mediante soluciones jabonosas. Por lo tanto, la protección parece sencilla: distancia social (siguiendo el acertado hashtag #YoMeQuedoEnCasa) y correcta higiene de manos.
Según un estudio reciente del Imperial College de Londres, si no se hubieran tomado las medidas de confinamiento el número de fallecimientos podría haber llegado a los 40 millones de personas en todo el mundo. Pero las medidas preventivas duraderas requieren la elaboración de vacunas eficaces. España tiene una larga tradición de investigación en virología reconocida internacionalmente, esencialmente concentrada, pero no únicamente, en Madrid y Barcelona.
Nuestros compañeros del Centro Nacional de Biotecnología del CSIC, los Drs. Luis Enjuanes y Mariano Esteban, están trabajando activamente para desarrollar vacunas frente al SARS-CoV-2 con dos métodos basados en principios distintos. El equipo de Enjuanes pretende atenuar el virus eliminando los genes de virulencia del SARS-CoV-2, manteniendo los genes responsables de la defensa inmunitaria.
Por su parte, el grupo de Mariano Estebanutiliza vectores virales para expresar la proteína S del virus en la superficie del virus de la vacuna de la viruela, que abre la puerta celular, esperando que desencadene la respuesta inmune. Otras estrategias de laboratorios públicos y empresas de varios países centran su esfuerzo en el uso de fragmentos del ARN viral, ADN o proteínas para generar esta respuesta protectora en el organismo.
Pero en la situación actual, obviamente, más que métodos preventivos se necesitan urgentemente los curativos, basados en compuestos antivirales que puedan tratar la infección. En pacientes infectados se están probando, entre otros antivirales, la (hidroxi)cloroquina, que impediría la ruta de entrada del virus, el Remdesivir, que actuaría sobre la replicasa viral, y la asociación Ritonavir/Lopinavir que inhibiría la proteasa usada por el virus para expresar su información.
Otros fármacos que se están administrando a los enfermos tienen el fin de controlar o modular la respuesta exagerada de la defensa inmune frente al propio virus, incluyendo interferones, corticosteroides, anticuerpos monoclonales como Tocilizumab frente al receptor de la interleuquina IL-6, Eculizumab inhibidor de la activación del complemento, etc.
Seamos optimistas, pero pacientes
Existen varios ensayos clínicos en curso a nivel mundial (algunos de ellos con participación activa de hospitales de nuestro país), pero por el momento los resultados no son concluyentes y los casos de éxito se refieren a ensayos a pequeña escala, sin validez estadística.
Hemos de ser optimistas, pero pacientes. En estas circunstancias se trata de una investigación bajo una gran presión y basada casi exclusivamente en el concepto de prueba y error con compuestos farmacológicamente aprobados y en ocasiones usados anteriormente frente a otros virus.
En la fase actual de socialización del miedo, por otra parte comprensible, es importante estar bien informados y no contribuir a transmitir, a velocidades más rápidas que la del propio virus, informaciones total o parcialmente falsas. Las segundas hacen aún más daño, porque introducen datos erróneos, manipulados y tendenciosos en un contexto aparentemente bien argumentado.
Esta pandemia debe servirnos para que definitivamente los gobiernos mundiales (en especial el español) se den cuenta de la imperiosa necesidad de invertir en la generación de conocimiento, y que la inversión en Ciencia, Educación y Sanidad deben ser la base sobre la que se construye una sociedad justa, igualitaria y próspera.
No olvidemos que, según un estudio de investigadores de la Escuela de Salud Pública de Boston publicado recientemente en la revista Science, aún en el caso de la eliminación aparente de la pandemia en los próximos meses, las estimaciones para los períodos pospandemia prevén rebrotes ocasionales hasta 2024. No nos queda otra opción que estar preparados.
La farmacéutica Mª Dolores García y el médico Juan García Costa también ha colaborado en este artículo.![]()
Albert Bosch, Catedrático de Microbiología. Dept. Genética, Microbiología y Estadística, Facultad de Biología, Universitat de Barcelona; Amelia Nieto, investigadora retirada, Centro Nacional de Biotecnología (CNB - CSIC); Ana María Doménech Gómez, Directora Departamento de Sanidad Animal, Universidad Complutense de Madrid; Carlos Briones, Científico Titular del CSIC y Vocal de la Junta Directiva de la Sociedad Española de Virología, Centro de Astrobiología (INTA-CSIC); Covadonga Alonso Martí, Profesor de Investigación, Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA); Inmaculada Casas Flecha, Investigadora del Centro Nacional de Microbiología, Instituto de Salud Carlos III; Javier Buesa Gómez, Catedrático de Microbiología, Universitat de València; Jesús Navas Castillo, Investigador Científico CSIC,. Profesor Asociado UMA, Instituto de Hortofruticultura Subtropical y Mediterránea "La Mayora" ( IHSM - CSIC) (EELM-CSIC) ; José Antonio López Guerrero, Profesor titular de Microbiología, Universidad Autónoma de Madrid; Josep Quer Sivila, Responsable de Recerca Bàsica del Virus de l’Hepatitis C del Laboratori de Malalties Hepàtiques Vall d’Hebron Institut de Recerca (VHIR), Hospital Universitario Vall d'Hebrón, Generalitat de Catalunya; Juan E. Echevarría Mayo, Investigador científico del Centro Nacional de Microbiología, Instituto de Salud Carlos III y Vicente Pallas Benet, Profesor de Investigación CSIC. Virología Molecular de Plantas, Instituto de Biología Molecular y Celular de Plantas (IBMCP - CSIC)
Este artículo fue publicado originalmente en The Conversation. Lea el original.