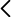El análisis genómico ha sido llevado a cabo por la Unidad Mixta en Infección y Salud Pública de la Universitat de València y la Fundación para el Fomento de la Investigación Sanitaria y Biomédica de la Comunidad Valenciana Fisabio, el Grupo de Investigación en Epidemiología Molecular, ambos liderados por Fernando González Candelas, catedrático de Genética e investigador del Instituto de Biología Integrativa de Sistemas (I2SysBio) de la Universitat de València y del CSIC, y el Servicio de Secuenciación y Bioinformática de Fisabio que coordinan Giuseppe d’Auria y Llúcia Martínez.
Los equipos de investigación valencianos han conseguido secuenciar el genoma completo de tres muestras de pacientes infectados por el COVID-19 procedentes del laboratorio de Microbiología del Hospital Clínico Universitario de Valencia, las primeras secuencias del virus SARS-CoV2 obtenidas en España. Esta secuenciación de genomas completos se suma al esfuerzo que se está haciendo a nivel mundial en todos los laboratorios para averiguar cuáles han sido las vías de transmisión y cómo se extienden los diferentes linajes del virus.
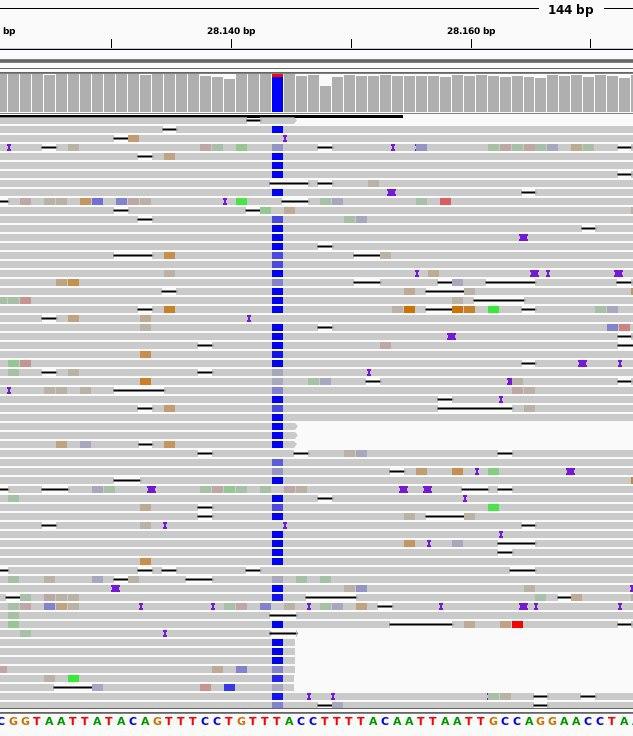
“El objetivo primordial es identificar con mayor fiabilidad los focos y las cadenas de transmisión del coronavirus. La principal conclusión del análisis de las primeras muestras es que las cepas proceden de rutas de transmisión diferentes”, explica el investigador Fernando González Candelas.
Las secuencias ya están accesibles en la base de datos de la Iniciativa GISAID, consorcio público dedicado al estudio del virus de la gripe (www.gisaid.org); la plataforma Nextstrain, que permite visualizar la progresión espacial y temporal de la pandemia a partir de los más de 500 genomas depositados desde el pasado diciembre por 40 países (https://nextstrain.org); y la base de datos Genbank de secuencias genéticas del NIH (National Institutes of Health de Estados Unidos) (https://www.ncbi.nlm.nih.gov/genbank/).
Los grupos formados por personal investigador especialista en virología, epidemiología y bioinformática han determinado que una de las cepas analizadas está relacionada con otras cepas europeas (de Italia, Alemania, Luxemburgo, Francia, Escocia, Países Bajos, etc.). “El siguiente paso será analizar secuencias de más muestras de pacientes de los hospitales de la Comunidad Valenciana para poder comprobar las vinculaciones entre ellas y con las cadenas de transmisión establecidas por el personal especialista en epidemiología”, explica Fernando González.
El análisis de genomas virales permite conocer las vías por las cuales ha entrado el virus a nuestra comunidad y cómo se transmite en estos momentos, lo que ayudará a las autoridades sanitarias a controlar mucho mejor la expansión del virus en nuestra comunidad. Además, la secuenciación del genoma del virus nos permite conocer las mutaciones que ha sufrido el virus desde que comenzó la epidemia y la conclusión a la que se ha llegado después del análisis realizado es que, hasta ahora, no se ha encontrado ninguna mutación asociada a una mayor virulencia, letalidad, o a alguna propiedad interesante desde el punto de vista clínico.
Las muestras han sido secuenciadas mediante MinIOn, un secuenciador de tercera generación de Oxford Nanopore Technologies. La infraestructura utilizada en la investigación ha sido posible gracias a la cofinanciación de la Unión Europea a través del Programa Operativo del Fondo Europeo de Desarrollo Regional (FEDER) de la Comunidad Valenciana 2014-2020.
Más información:
http://fisabio.san.gva.es/ca/inicio
Pies de fotografía:
1. Grupo del equipo de secuenciación
2. Imagen de un fragmento de las secuencias del virus SARS-CoV-2 analizado. En azul se observa una mutación del virus respecto al genoma de referencia.
3. Filogenia del coronavirus SARS-CoV-2. Las flechas señalan los linajes de los virus aislados en Valencia.
Imágenes:
.jpg)