Programa de Biología Teórica y Computacional
Los animales marinos de cuerpo blando, como los nudibranquios, utilizan compuestos bioactivos como defensa contra depredadores. Anteriormente, en nuestro trabajo sobre el nudibranquio Doriopsilla fulva hemos descubierto que estos compuestos pueden ser producidos por simbiontes bacterianos en lugar de los propios animales. Estas bacterias simbióticas representan nuevos linajes bacterianos no cultivables que producen moléculas bioactivas. El proyecto Nudi-Symb busca explorar más especies de nudibranquios. Utilizaremos técnicas novedosas para estudiar células individuales bacterianas, secuenciaremos el genoma de nudibranquio y utilizaremos transcriptómica para entender la interacción entre estos animales y sus bacterias simbióticas, con el objetivo de contribuir al desarrollo de nuevos fármacos médicos.
El consorcio FRAILTECH busca desarrollar una tecnología de detección precoz del síndrome de fragilidad basada en arrays de ADN. Se combinarán técnicas de secuenciación de ADN y análisis metagenómicos para crear un conjunto de datos que permita evaluar el riesgo de fragilidad. Además, se validará una calculadora de riesgo multiescalar que integrará información genética, de microbiota y variables clínicas. El proyecto también busca generar bases de datos para la comunidad de investigación de fragilidad.
En este estudio el objetivo es el desarrollo de una solución rápida y económica para detectar el riesgo de deterioro en las extremidades inferiores en personas mayores. Utilizando tecnologías de secuenciación de ADN y análisis metagenómico, se creará un panel de referencia que incluirá datos genéticos y microbióticos así como una calculadora de riesgo y una base de datos para investigaciones relacionadas.
El proyecto LIVERGUT busca evaluar los efectos del trasplante de microbioma fecal (FMT) en pacientes con cirrosis. Dos ensayos aleatorizados de fase III serán realizados para evaluar la reducción de la hipertensión portal y las complicaciones de la cirrosis.
Los organismos multicelulares tienen una relación simbiótica con microbios, y esta asociación es esencial para su funcionamiento. Los microbiomas, que son comunidades de microbios, pueden clasificarse en tipos específicos. Esto es relevante en el ámbito clínico, ya que ayuda en el diagnóstico, identificación de factores de riesgo y predicción del éxito del tratamiento. CLOM consiste en una base de datos de perfiles de microbiomas y un clasificador de perfiles que emplea técnicas de agrupación no supervisadas. El principal objetivo es permitir la caracterización y predicción de la microbiota y su relación con enfermedades, así como para la asignación de nuevas muestras a tipos de comunidades microbianas asociadas a enfermedades.
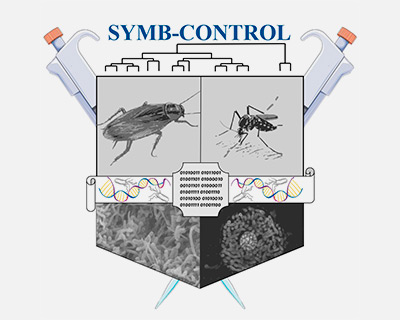
El proyecto se centra en el estudio de las interacciones entre bacteriófagos y bacterias en la microbiota de la cucaracha Blattella germanica. Se hipotetiza que los bacteriófagos afectan la diversidad bacteriana y que la respuesta del huésped al estrés, como los antibióticos o las temperaturas extremas, altera estas interacciones y el metabolismo del huésped.
Cambios con la edad de las interacciones de la microbiota con su hospedador humano y determinación de un núcleo permanente de simbiontes mutualistas.


 Nudi-Symb
Nudi-Symb
 FRAILTECH
FRAILTECH
 LEGTECH
LEGTECH
 LIVERGUT
LIVERGUT
 CLOM
CLOM
 TEANS-PB
TEANS-PB
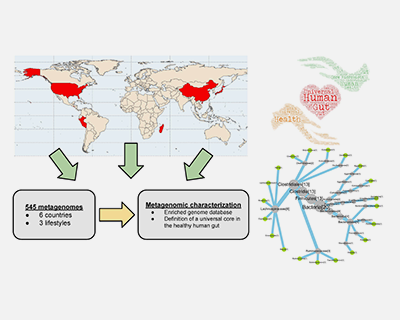 MUTUALCORE
MUTUALCORE
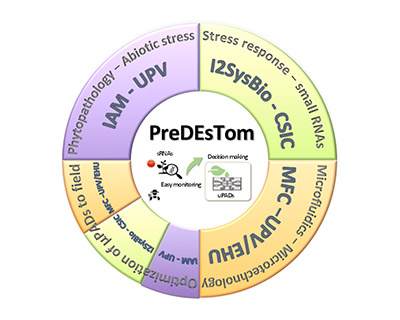 PreDeSTom
PreDeSTom
 BotVidSENSOR
BotVidSENSOR
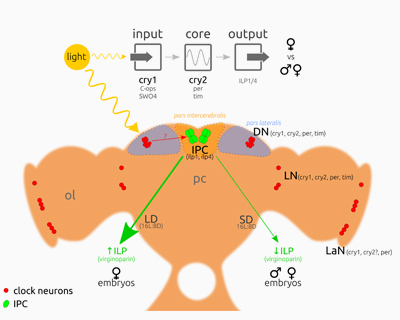 DIMITRA
DIMITRA
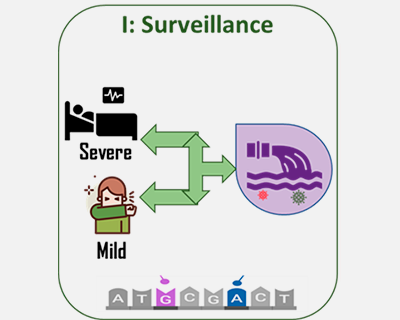 EvolCovid
EvolCovid
 Biomarcadores de patogenicidad en Legionella spp
Biomarcadores de patogenicidad en Legionella spp
 ExOHAR
ExOHAR
 SpaceWorms
SpaceWorms
 mol-TB-host
mol-TB-host
 INVEST/2023/173
INVEST/2023/173
 HMW-nudibranch
HMW-nudibranch
 HN-OMICS
HN-OMICS
 SYMB-CONTROL
SYMB-CONTROL
 CDEIGENT
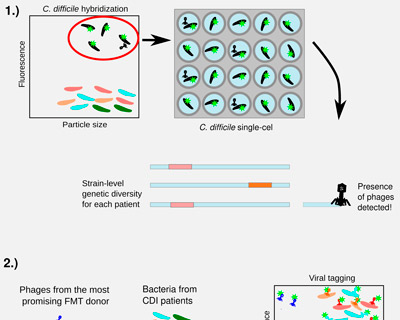
CDEIGENT
 exRNA-PATH
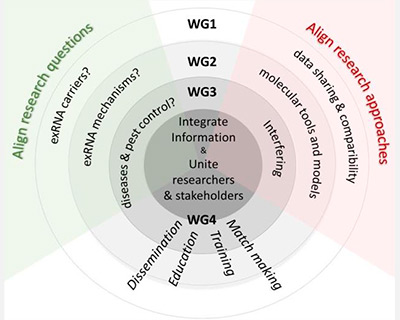
exRNA-PATH
 COALESCE
COALESCE
 EVOPUNCH
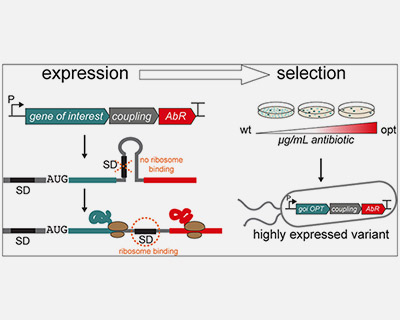
EVOPUNCH
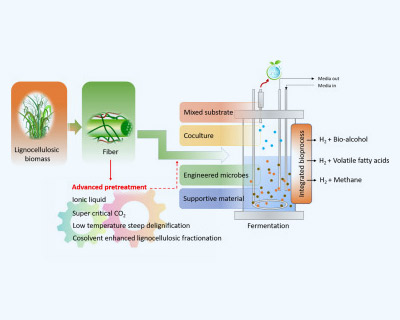 ProtoSynH2
ProtoSynH2
 MICRO4BIOGAS
MICRO4BIOGAS
 SINNBIOSIS
SINNBIOSIS